[解決済み] 加重ロジスティック回帰 (R)
2022-02-14 22:43:31
質問
成功者の割合とサンプルサイズ、独立変数のサンプルデータがある場合、Rでロジスティック回帰を試みているところです。
次のコードは、私が望むことを行い、賢明な結果を与えるように見えますが、賢明なアプローチとは思えません。
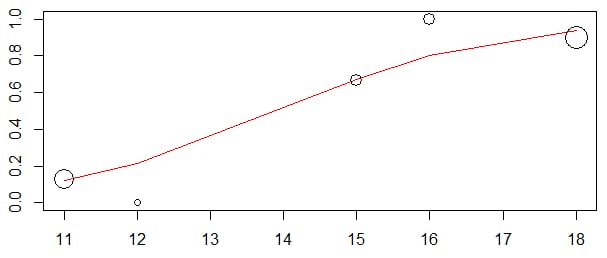
datf <- data.frame(prop = c(0.125, 0, 0.667, 1, 0.9),
cases = c(8, 1, 3, 3, 10),
x = c(11, 12, 15, 16, 18))
datf2 <- rbind(datf,datf)
datf2$success <- rep(c(1, 0), each=nrow(datf))
datf2$cases <- round(datf2$cases*ifelse(datf2$success,datf2$prop,1-datf2$prop))
fit2 <- glm(success ~ x, weight=cases, data=datf2, family="binomial")
datf$proppredicted <- 1 / (1 + exp(-predict(fit2, datf)))
plot(datf$x, datf$proppredicted, type="l", col="red", ylim=c(0,1))
points(datf$x, datf$prop, cex=sqrt(datf$cases))
というのは、それなりに理にかなっているように見えます。
しかし、私が不満なのは
datf2
を、データを重複させて成功・失敗を分ける方法として このようなものは必要でしょうか?
あまりない質問ですが、予測比率の計算をもっときれいにする方法はないでしょうか?
解決方法は?
そのような人工的なデータを構築する必要はありません。
glm
は、与えられたデータセットからモデルを適合させることができます。
> glm(prop ~ x, family=binomial, data=datf, weights=cases)
Call: glm(formula = prop ~ x, family = binomial, data = datf, weights = cases)
Coefficients:
(Intercept) x
-9.3533 0.6714
Degrees of Freedom: 4 Total (i.e. Null); 3 Residual
Null Deviance: 17.3
Residual Deviance: 2.043 AIC: 11.43
非整数の #successes" に関する警告が表示されますが、これは次の理由からです。
glm
がバカになっています。構築したデータセットのモデルと比較してみてください。
> fit2
Call: glm(formula = success ~ x, family = "binomial", data = datf2,
weights = cases)
Coefficients:
(Intercept) x
-9.3532 0.6713
Degrees of Freedom: 7 Total (i.e. Null); 6 Residual
Null Deviance: 33.65
Residual Deviance: 18.39 AIC: 22.39
回帰係数(したがって予測値)は基本的に等しいです。しかし、人工的なデータポイントを作成したため、残差devianceとAICは疑わしいです。
関連
-
[解決済み] 変数の型(リスト)が無効です
-
[解決済み】scale_color_manual()が動作しない件
-
[解決済み】lis[[i]]のエラー:1つ未満の要素を選択しようとした場合
-
[解決済み】 boxplotに色を追加する - "離散的なスケールに連続的な値が供給される "エラー
-
[解決済み】 file(filename, "r", encoding = encoding) : cannot open the connectionでエラーが発生する。
-
[解決済み】Rで文字ベクトルから引用符を削除する
-
[解決済み】r Error dim(X) must have a positive length?
-
[解決済み】R: predict() の数値 'envir' 引数が長さ1でない。)
-
[解決済み】Rでデータフレームのサブセットをプロットする方法は?
-
[解決済み】Rで、Error: ggplot2 doesn't know how to handle of data of class numericに対処する。
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
[解決済み】R:関数に有限な'ylim'値が必要
-
[解決済み】エラー。Rの次元数が正しくない
-
[解決済み] 変数の型(リスト)が無効です
-
[解決済み】library(ggplot2)でエラー:'ggplot2'というパッケージは存在しません。
-
[解決済み】lis[[i]]のエラー:1つ未満の要素を選択しようとした場合
-
[解決済み】GLM解析での警告
-
[解決済み】「'dimnames' [1]の長さが配列の範囲と等しくない」とはどういう意味ですか?
-
[解決済み] lmer エラー: グループ化係数は観測数未満でなければならない
-
[解決済み] [Solved] read.csv warning 'EOF within quoted string' prevents complete reading of file.
-
[解決済み】forループを実行すると「要因のレベルセットが異なる」というエラーが発生する