[解決済み] ggplot2 geom_line で色を手動で指定する
2022-03-10 07:29:09
質問
以下のようなグラフを作成しようとしているのですが、手動で色を指定したいのです。
同じ Bgrnd_All に属する遺伝子型が複数あるので、遺伝子型ごとにプロットしたいのですが、プロットした線に別々に表示させたいのですが。
しかし、私はBgrnd_Allによって線に色を付けたいのですが、具体的にはscale_fill_manualで使用する順序/色で行います。
このとき、scale_fill_manual の値は geom_line で定義された既存の色を上書きしません。どうすればよいのでしょうか?
ご指摘いただけるとありがたいです。
[下記グラフのデータ][1]。 https://www.dropbox.com/s/9nmu87wkh2yqfxn/summary_200_exp2.csv?dl=0
pd <- position_dodge(1)
ggplot(data=summary.200.exp2, aes(x=Time, y=Length, colour=Genotype, group=Genotype)) +
geom_errorbar(aes(ymin=Length - se, ymax=Length + se), colour="black", width=1, position=pd) +
geom_line(aes(colour=Bgrnd_All), position=pd, size =1) +
scale_x_continuous(breaks=c(0,17,22,41,89)) + #using breaks of when sampled
scale_fill_manual(values=c(Avalon="#000066",Av_A="#663399",Av_B="#339999",Cadenza="CC0033",Cad_A="FF6600",Cad_B="FF9933"))+
ylab("leaf segment width (mm)") +
xlab("Time") +
theme(axis.title = element_text(size=14,face="bold"),
axis.text = element_text(size=14),
strip.text.y = element_text(size=14))
A
dput
のデータです。
summary.200.exp2 <- structure(list(X = 1:40,
Genotype = structure(c(1L, 1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L, 3L, 3L, 4L, 4L, 4L, 4L, 4L, 5L, 5L, 5L, 5L, 5L, 6L, 6L, 6L, 6L, 6L, 7L, 7L, 7L, 7L, 7L, 8L, 8L, 8L, 8L, 8L), .Label = c("4.18", "4.41", "7.50", "7.59", "8.51", "8.77", "Avalon", "Cadenza"), class = "factor"),
Time = c(0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L, 0L, 17L, 22L, 41L, 89L),
Bgrnd_All = structure(c(4L, 4L, 4L, 4L, 4L, 5L, 5L, 5L, 5L, 5L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 2L, 2L, 2L, 5L, 5L, 5L, 5L, 5L, 4L, 4L, 4L, 4L, 4L, 3L, 3L, 3L, 3L, 3L, 6L, 6L, 6L, 6L, 6L), .Label = c("Av_A", "Av_B", "Avalon", "Cad_A", "Cad_B", "Cadenza"), class = "factor"),
N = c(43L, 48L, 44L, 47L, 48L, 22L, 21L, 26L, 27L, 25L, 36L, 24L, 44L, 48L, 45L, 50L, 26L, 52L, 54L, 53L, 38L, 52L, 52L, 49L, 50L, 39L, 39L, 42L, 38L, 42L, 84L, 42L, 84L, 42L, 42L, 50L, 26L, 53L, 27L, 27L),
Length = c(1.17423255813953, 1.58852083333333, 1.71263636363636, 1.86736170212766, 2.0331875, 1.07563636363636, 1.49866666666667, 1.48734615384615, 1.66796296296296, 2.15416, 1.08716666666667, 1.09858333333333, 1.24593181818182, 1.30827083333333, 1.81537777777778, 1.15672, 1.8475, 1.96815384615385, 2.01822222222222, 2.5057358490566, 1.14697368421053, 1.40276923076923, 1.49832692307692, 1.76981632653061, 2.27954, 1.18312820512821, 1.75928205128205, 1.86195238095238, 1.91426315789474, 2.26883333333333, 1.10839285714286, 1.97902380952381, 2.03271428571429, 2.15685714285714, 2.8227380952381, 1.08658, 1.68880769230769, 1.7277358490566, 1.9232962962963, 2.13466666666667),
sd = c(0.218740641945063, 0.357307960001092, 0.377931031662453, 0.416137123383518, 0.440003996899158, 0.176915784499843, 0.426273190962478, 0.305677731254037, 0.450036449932454, 0.48642939535627, 0.15212823538055, 0.175160775008132, 0.293836087650785, 0.282464815326021, 0.346608194369436, 0.211422397593258, 0.408328617659845, 0.413460118977535, 0.419730221832425, 0.508692484972064, 0.217587942685885, 0.207510416973071, 0.245473270071832, 0.377310585673427, 0.536134471785516, 0.159925670150259, 0.298319411009668, 0.338847829173593, 0.296186727462412, 0.445638589029855, 0.162594700328365, 0.308723610551514, 0.318831396748337, 0.381781291715339, 0.402059458017902, 0.167826451905484, 0.257140275994371, 0.338637947743116, 0.362428434825926, 0.343680867174096),
se = c(0.0333576351702583, 0.0515729617225566, 0.0569752467571038, 0.0606998379642952, 0.06350910651356, 0.0377185719899813, 0.0930204363959963, 0.0599483352513503, 0.0866095551712153, 0.097285879071254, 0.0253547058967583, 0.0357545434766975, 0.0442974569365289, 0.040770284291269, 0.0516692989445678, 0.0298996422065822, 0.0800798303617661, 0.0573366022820362, 0.0571180485063685, 0.0698742866122227, 0.0352974252834232, 0.0287765172534354, 0.0340410177692235, 0.053901512239061, 0.0758208641254813, 0.0256086023072023, 0.0477693365291991, 0.052285355168868, 0.0480478318490224, 0.0687635271596866, 0.0177405362346046, 0.0476370873204908, 0.0347873573697084, 0.0589101322645314, 0.0620391212561054, 0.0237342444409691, 0.0504293571163821, 0.046515499476421, 0.0697493848029077, 0.0661414137260961),
ci = c(0.0673184331863912, 0.103751416510302, 0.114901535684132, 0.122182436693452, 0.127763842564108, 0.0784400645137227, 0.194037230170767, 0.123465907623535, 0.178028490322197, 0.200788185881879, 0.0514727894594648, 0.0739639084701291, 0.0893343358495282, 0.0820192326650262, 0.104132629687123, 0.0600855805773719, 0.164927497928001, 0.11510803218647, 0.11456429705202, 0.140213013986381, 0.0715193770736051, 0.0577712690042106, 0.0683401947985261, 0.108376253996364, 0.152367731004308, 0.0518419050566429, 0.0967039660836575, 0.105592416917608, 0.0973541547573791, 0.138870760371045, 0.0352852130493688, 0.0962050495562246, 0.06919065466693, 0.118971425682342, 0.125290547146885, 0.0476957499005439, 0.103861205171753, 0.0933401784102089, 0.143371913789607, 0.135955623027448)),
.Names = c("X", "Genotype", "Time", "Bgrnd_All", "N", "Length", "sd", "se", "ci"), class = "data.frame", row.names = c(NA, -40L))
解決方法は?
コメントで@jubaさんが述べられているように
scale_colour_manual
の代わりに
scale_fill_manual
. さらに、あなたは1つのプロットに多くの線とエラーバーを描こうとしています。それらは互いに重なりすぎていて、そのため線/エラーバーを区別するのが難しいです。
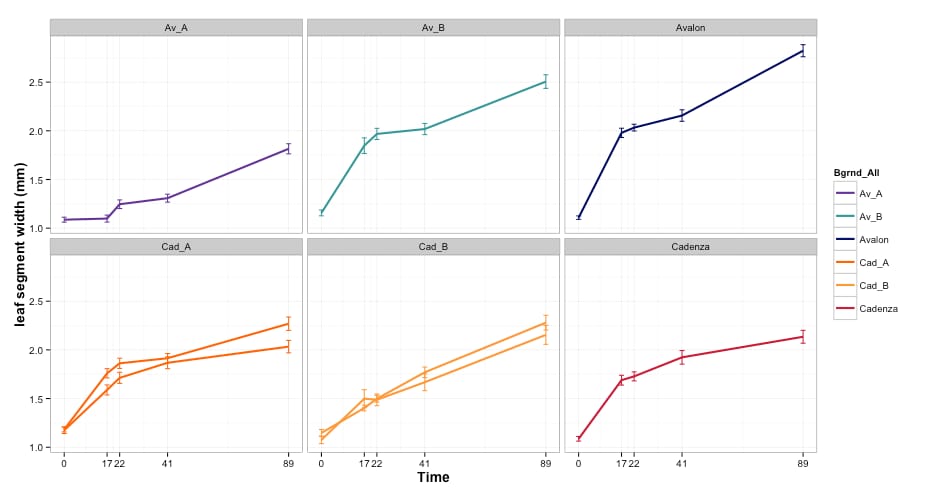
ファセットを使用した例(とコードの簡略化)。
ggplot(summary.200.exp2, aes(x=Time, y=Length, group=Genotype)) +
geom_line(aes(colour=Bgrnd_All), size =1) +
geom_errorbar(aes(ymin=Length-se, ymax=Length+se, colour=Bgrnd_All), width=2) +
scale_x_continuous("Time", breaks=c(0,17,22,41,89)) +
scale_colour_manual(values=c(Avalon="#000066",Av_A="#663399",Av_B="#339999",Cadenza="#CC0033",Cad_A="#FF6600",Cad_B="#FF9933"))+
ylab("leaf segment width (mm)") +
theme_bw() +
theme(axis.title = element_text(size=14,face="bold"), axis.text = element_text(size=10)) +
facet_wrap(~Bgrnd_All, ncol=3)
というのがあります。
関連
-
[解決済み】基本 - T-検定 -> グループ化因子は正確に2水準でなければならない
-
[解決済み] 変数の型(リスト)が無効です
-
[解決済み】長いオブジェクトの長さは、短いオブジェクトの長さの倍数ではない?[重複]。
-
[解決済み] 因子を日付形式に変換するにはどうすればいいですか?
-
[解決済み】Rで結果の行数がベクトル長(arg 2)の倍数でない件
-
[解決済み】各グループの上位3要素を計算する際に「数値型、論理型、複合型でのみ可能な演算」を回避する方法
-
[解決済み】起動時にパーをデフォルト値にリセットする方法
-
[解決済み】Rで「中断されたプロミスの評価を再開する」という警告を回避する
-
[解決済み] ggplot2 の軸ラベルを回転させ、間隔を空ける
-
[解決済み】ggplot2によるサイド・バイ・サイド・プロット
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
[解決済み】R:関数に有限な'ylim'値が必要
-
[解決済み] 不適合な配列のコードエラー
-
[解決済み】'builtin'型のオブジェクトはsubsetableではない【重複
-
[解決済み】エラー。Rの次元数が正しくない
-
[解決済み] Stataバージョン5-12の.dtaファイルではない
-
[解決済み] write.tableしようとすると、未実装の型リストが表示される。
-
[解決済み】 if/while (条件) {: TRUE/FALSEが必要な場所に値がない場合のエラー
-
[解決済み】行列式で「数値/複雑な行列/ベクトルの引数を必要とする」というエラーが発生する?
-
[解決済み】Rで相対的な頻度をプロットするためにhistをどのように使用しますか?
-
[解決済み】Rで「中断されたプロミスの評価を再開する」という警告を回避する