[解決済み] TukeyHSDによる誤差
2022-02-18 04:15:24
質問
以下のようなデータがあり、ANOVAを実行してすべての列間の差をチェックしようとします。どの程度有意に違うのか等。
df<- structure(list(color = structure(c(3L, 4L, 3L, 4L, 4L, 4L, 4L,
4L, 4L, 4L), .Label = c("B", "G", "R", "W"), class = "factor"),
type = 1:10, X1 = c(0.006605138, 0.001165448, 0.006975109,
0.002207839, 0.00187902, 0.002208638, 0.001199808, 0.001162252,
0.001338847, 0.001106317), X2 = c(0.006041392, 0.001639298,
0.006140877, 0.002958169, 0.002744017, 0.003107995, 0.001729594,
0.001582564, 0.001971713, 0.001693236), X3 = c(0.024180351,
0.002189061, 0.027377442, 0.002886651, 0.002816333, 0.003527908,
0.00231891, 0.001695633, 0.00212034, 0.001962923)), .Names = c("color",
"type", "X1", "X2", "X3"), row.names = c(NA, 10L), class = "data.frame")
まず、次のコマンドでANOVAを実行します。
anovar= aov(type~.,df)
を実行し、次のように出力をまとめる。
summary(anovar)
ここまでは順調で、うまく動作しています。しかし、TukeyHSDを実行しようとすると、構造に問題があるようです。以下のようなエラーになります。検索してみましたが、同様の状況は見つかりませんでした。何かコメントがあればお願いします。
TukeyHSD(anovar)
# Error in rep.int(n, length(means)) : unimplemented type 'NULL' in 'rep3'
# In addition: Warning messages:
# 1: In replications(paste("~", xx), data = mf) : non-factors ignored: X1
# 2: In replications(paste("~", xx), data = mf) : non-factors ignored: X2
# 3: In replications(paste("~", xx), data = mf) : non-factors ignored: X3
解決方法は?
TukeyHSD のドキュメントの説明にあるように,この関数は,あるレベルの平均値の差に対する信頼区間のセットを作成します. 因子 を、指定された族単位の網羅確率で指定する。
つまり、実行するには、データセットにファクターが含まれている必要があります。そこで、以下のように因子を選択すると動作します。
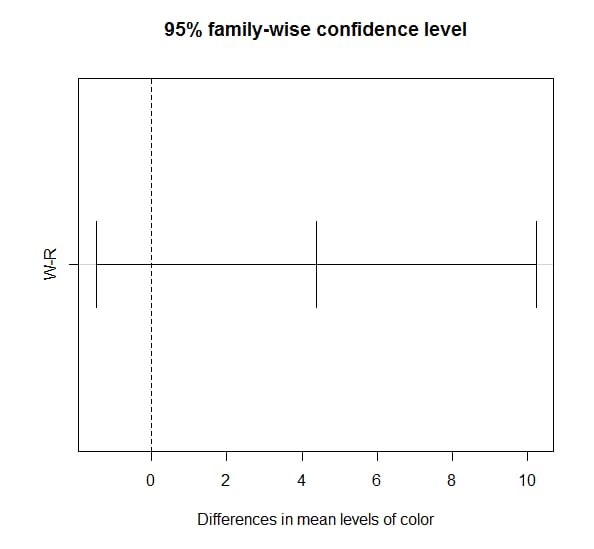
> TukeyHSD(anovar, which = 'color') #color is the only categorical data
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = type ~ ., data = df)
$color
diff lwr upr p adj
W-R 4.375 -1.465325 10.21532 0.1121168
また、非因子X1, X2, X3は無視されるという警告が表示されます。
TukeyHSD オブジェクトを印刷するには、それを保存して、次のようにします。
plot
. このように
plot
メソッド(および
print
メソッド) を使用しています。
forplot <- TukeyHSD(anovar, which = 'color')
plot(forplot)
関連
-
[解決済み】R: 複数行の ggplot2 コマンドで「単項演算子エラー」が発生する。
-
[解決済み】model.frame.defaultでのエラー:変数の長さが異なる
-
[解決済み】R - if文の引数の長さが0である。
-
[解決済み】値を置き換えようとすると、"データフレームの添え字の割り当てで欠損値は許可されない"
-
[解決済み] テスト
-
[解決済み】reshape2 meltの警告メッセージ
-
[解決済み】エラー。Rの'break'の数が無効
-
[解決済み】ggplotのエラー。関数型オブジェクトのスケールを自動的に選択する方法がわかりません。
-
[解決済み】Rの整数オーバーフローとは何ですか、そしてどのように起こるのですか?
-
[解決済み】R: predict() の数値 'envir' 引数が長さ1でない。)
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
[解決済み】"置換する項目数が置換長の倍数でない "と表示されるのはなぜですか?
-
[解決済み】R:関数に有限な'ylim'値が必要
-
[解決済み】エラー:'dimnames' [2]の長さが配列の範囲と等しくない [終了しました]
-
[解決済み】エラー:私のコードで予期しないシンボル/入力/文字列定数/数値定数/SPECIALが発生した
-
[解決済み】 'height' はベクトルか行列でなければならない barplot のエラー
-
[解決済み】RでのMLEエラー:'vmmin'の初期値が有限でない
-
[解決済み】データのマージ - fix.by(by.x, x)のエラー)
-
[解決済み】Rのメモリ管理/サイズn Mbのベクトルを割り当てられない
-
[解決済み】R Markdown - html出力でフォントサイズとフォントタイプを変更する
-
[解決済み】.External.graphics Rでエラーが発生しました。