R ヒートマップ描画の学習ノート

taoyanです。 R言語中国語コミュニティ寄稿者、疑似コーダー、R言語愛好家、オープンソースが大好き。
<スパン 個人ブログ:https://ytlogos.github.io/

<スパン はじめに
この記事では、以下のRパッケージと関数を用いて、静的および対話的なヒートマップをプロットします。
heatmap(): 単純なヒートマップを描画するための関数
heatmap.2():拡張ヒートマップを描画する関数です。
d3heatmap:対話的ヒートマップ描画のためのRパッケージ
ComplexHeatmap:R&bioconductor パッケージ - 複雑なヒートマップのプロット、アノテーション、アラインメント (ゲノムデータ解析に最適)
<スパン データ準備
Rの組み込みデータセットmtcarsを使用する
<スパン df <- as.matrix((scale(mtcars)))# 正規化、行列化
<スパン 基本的な関数を使ったシンプルなヒートマップ
主な関数はheatmap(x, scale="row") です。
x:データマトリックス
scale: 異なる方向を示す、オプションの値: row, columa, none
デフォルトのplotheatmap(df, scale = "none")です。
<スパン カスタムカラーを使用する
col <- colorRampPalette(c("red", "white", "blue"))(256)。
ヒートマップ(df, scale = "none", col=col)
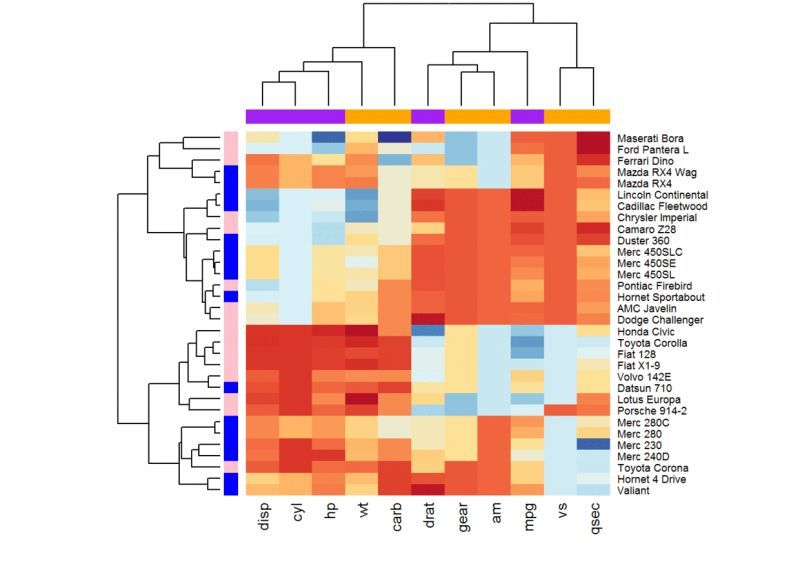
<ブロッククオート#RColorBrewer のカラーパレット名を使用します。
library(RColorBrewer)col <- colorRampPalette(brewer.pal(10, "RdYlBu"))(256)# 自己設定パレット dim(df)# 表示行数
## [1] 32 11
heatmap(df, scale = "none", col=col, RowSideColors = rep(c("blue", "pink"), each=16))です。
ColSideColors = c(rep("purple", 5), rep("orange", 6)))。
#パラメータRowSideColorsとColSideColorsは、それぞれ行と列の色などを注釈するために使われ、help(heatmap)の詳細を示します。
<スパン ヒートマップの充実
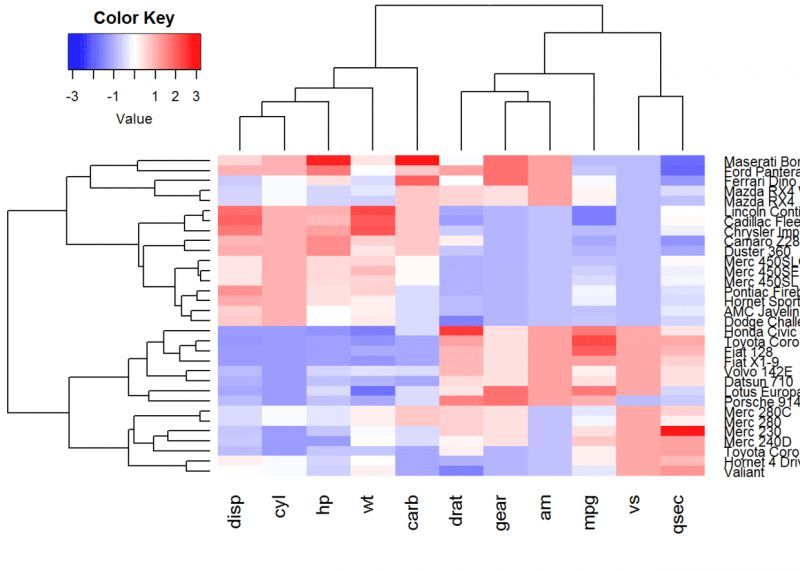
関数 heatmap.2()
ヒートマッププロットにおける多くの拡張機能を提供します。この関数は gplots パッケージでラップされています。
library(gplots)heatmap.2(df, scale = "none", col=bluered(100)),
trace = "none", density.info = "none")# help(heatmap.2()) にあるように、他にもパラメータがあるようです。
<スパン
インタラクティブなヒートマップ描画
d3heatmapパッケージは、インタラクティブなヒートマッププロットを生成するために使用することができます。
if (!require("devtools"))
install.packages("devtools")をインストールします。
devtools::install_github("rstudio/d3heatmap")
d3heatmap()関数は、以下の関数でインタラクティブなヒートマップを作成するために使用されます。
<スパン 1. 興味のあるヒートマップセルにマウスオーバーすると、行名と対応する値が表示されます。
2. 領域を選択して拡大・縮小することができます
<ブロッククオートlibrary(d3heatmap)d3heatmap(df, colors = "RdBu", k_row = 4, k_col = 2)
k_row, k_col は、それぞれ行と列のツリーマップの枝を着色するのに必要なグループの数を指定します。詳細な情報は help(d3heatmap()) から得ることができます。
<スパン dendextendパッケージによるヒートマップの強化
dendextend パッケージは、他のパッケージの機能を拡張するために使用することができます。
library(dendextend)# 行の並び順
<スパン Rowv <- mtcars %>% scale %>% dist %>%。
hclust %>% as.dendrogram %>%.
set("branches_k_color", k = 3) %>%.
set("branches_lwd", 1.2) %>% ladderize# columns# の順番を指定します。
データを転置する必要があります
Colv <- mtcars %>% scale %>% t %>% dist>%.
hclust %>% as.dendrogram %>%.
set("branches_k_color", k = 2, value = c("orange", "blue")) %>% set("branches_lwd", 1.2) %>% ladderize
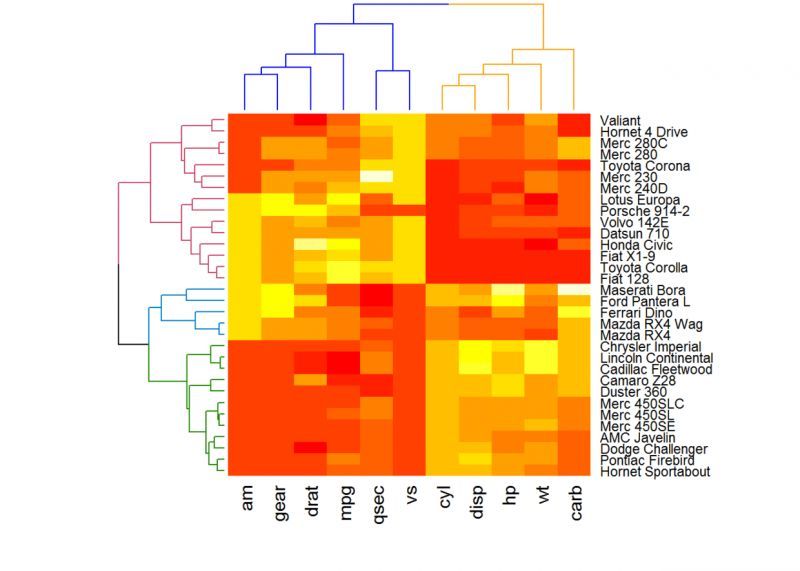
heatmap()関数の強化
---
heatmap(df, Rowv = Rowv, Colv = Colv, scale = "none")
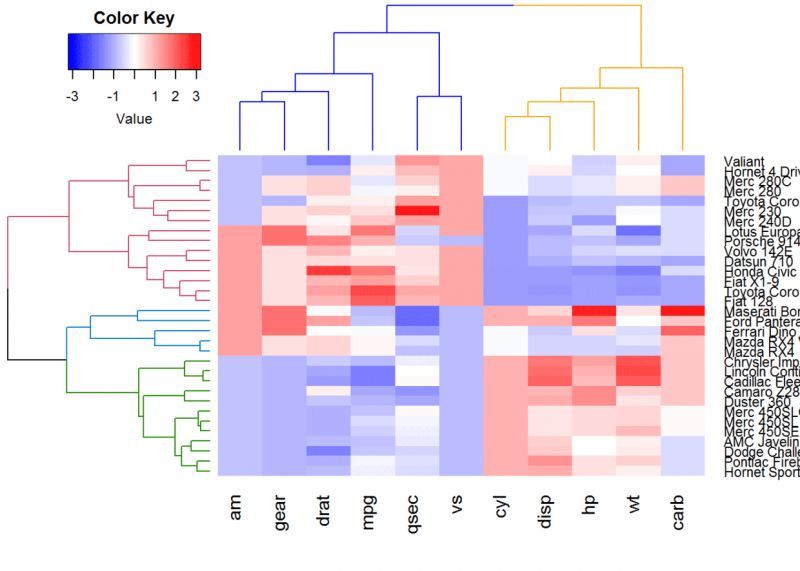
#heatmap.2()関数を強化する
heatmap.2(df, scale = "none", col = bluered(100), Rowv = Rowv, Colv = Colv, trace = "none", density.info = "none")
#インタラクティブな描画機能を強化
d2heatmap()d3heatmap(scale(mtcars), colors = "RdBu", Rowv = Rowv, Colv = Colv)
<スパン
複雑なヒートマップの作図
ComplexHeatmap パッケージは、複雑なヒートマップを描くための bioconductor パッケージで、複数のヒートマップの配置と注釈を柔軟に行うことができます。また、異なるソースからの異なるデータ間の関連性をヒートマップで可視化することができます。以下のコードでインストールできます。
<ブロッククオートif (!require("devtools")) install.packages("devtools")
devtools::install_github("jokergoo/ComplexHeatmap")
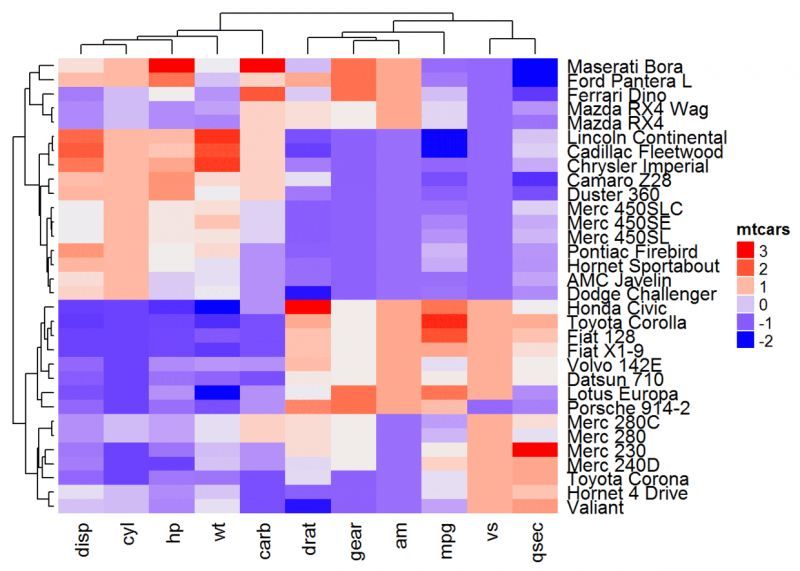
ComplexHeatmapパッケージのメイン関数はHeatmap()で、形式は以下の通りです。Heatmap(matrix, col, name) の形式です.
matrix: マトリックス
col: カラーベクトル(離散的なカラーマッピング)またはカラーマッピング関数(行列が連続数である場合).
name: ヒートマップの名前
library(ComplexHeatmap)
ヒートマップ(df, name = "mtcars")
#セルフセッティング・カラー
ライブラリ(circlize)
ヒートマップ(df, name = "mtcars", col = colorRamp2(c(-2, 0, 2), c("green", "white", "red")) ))
<スパン カラーパレットを使用する <スパン
ヒートマップ(df, name = "mtcars",col = colorRamp2(c(-2, 0, 2), brewer.pal(n=3, name="RdBu")))
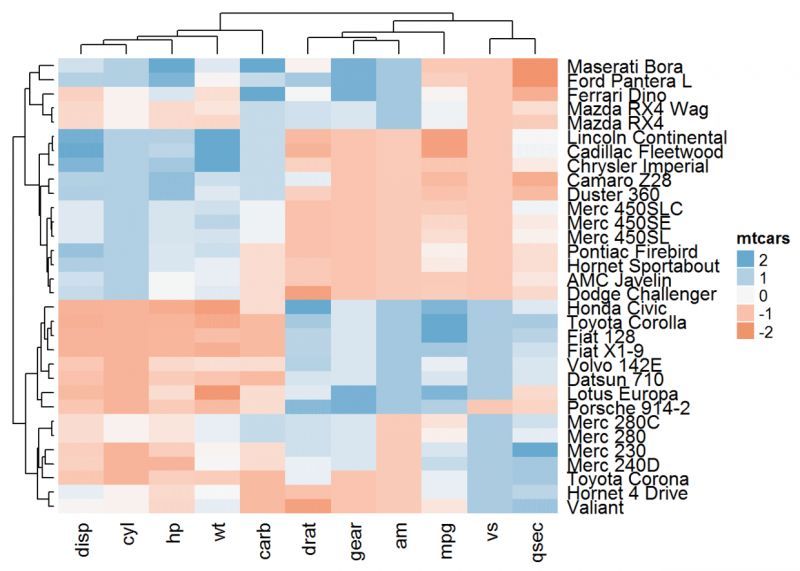 <スパン
<スパン
#カスタムカラー
mycol <- colorRamp2(c(-2, 0, 2), c("blue", "white", "red")))
#ヒートマップおよび行のタイトルの設定
Heatmap(df, name = "mtcars", col = mycol, column_title = "列タイトル", row_title =
行のタイトル")
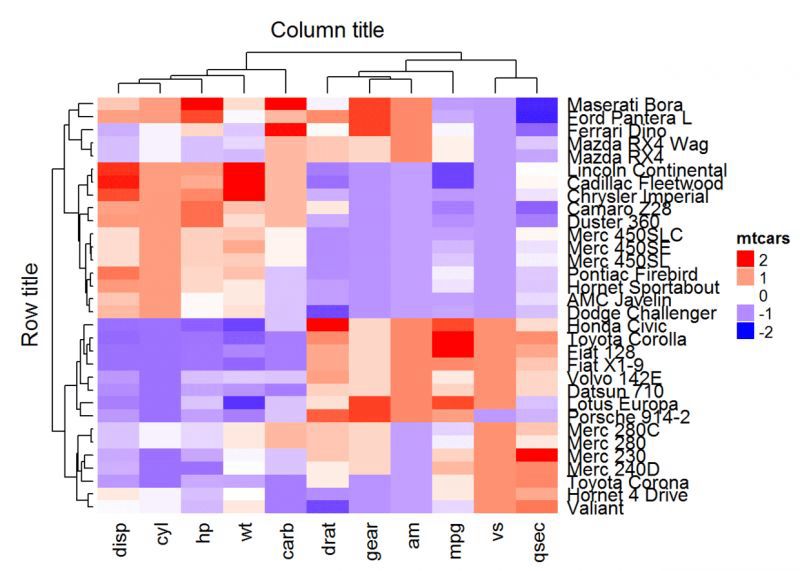
行ヘッダーのデフォルト位置は "left"、列ヘッダーのデフォルト位置は "top" であることに留意してください。これは、以下のオプションで変更することができます。
row_title_side: 許容される値は "left" または "right" です (例: row_title_side = "right").
column_title_side: 許容値は "top" または "bottom" (例: colum3, n_title_side = "bottom ") フォントとサイズは、以下のオプションで変更することもできます。
row_title_gp: 行のテキストを描画するためのグラフィックパラメータです。
column_title_gp: 列のテキストをプロットするためのグラフィカルパラメータ。
Heatmap(df, name = "mtcars", col = mycol, column_title = "Column title"。
column_title_gp = gpar(fontsize = 14, fontface = "bold"),
row_title = "行のタイトル", row_title_gp = gpar(fontsize = 14, fontface = "bold")))

上のRのコードでは、fontfaceに指定できる値は整数か文字列です。1 = plain, 2 = bold, 3 = italic, 4 = bold italicです。文字列の場合、有効な値は次のとおりです。
プレーン、ボールド、イタリック、オブリーク、ボールド.イタリックがあります。
行/列の名前を表示する。
show_row_names: 行の名前を表示するかどうか。デフォルトは TRUE。
show_column_names: カラム名を表示するかどうか。デフォルトは TRUE。
<ブロッククオートヒートマップ(df, name = "mtcars", show_row_names = FALSE)
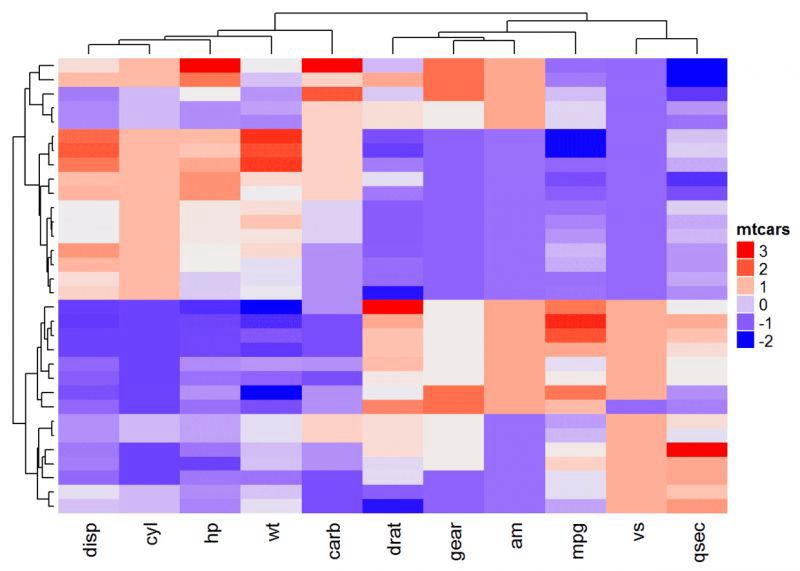
<スパン クラスターの外観を変更する
デフォルトでは、行と列がクラスタに含まれます。これはパラメータを使って変更することができます。
cluster_rows = FALSE. TRUE の場合、行にクラスターを作成します。
cluster_columns = FALSE. TRUE の場合、クラスタにカラムを配置します。
# 行のクラスタを非活性化する
ヒートマップ(df, name = "mtcars", col = mycol, cluster_rows = FALSE)
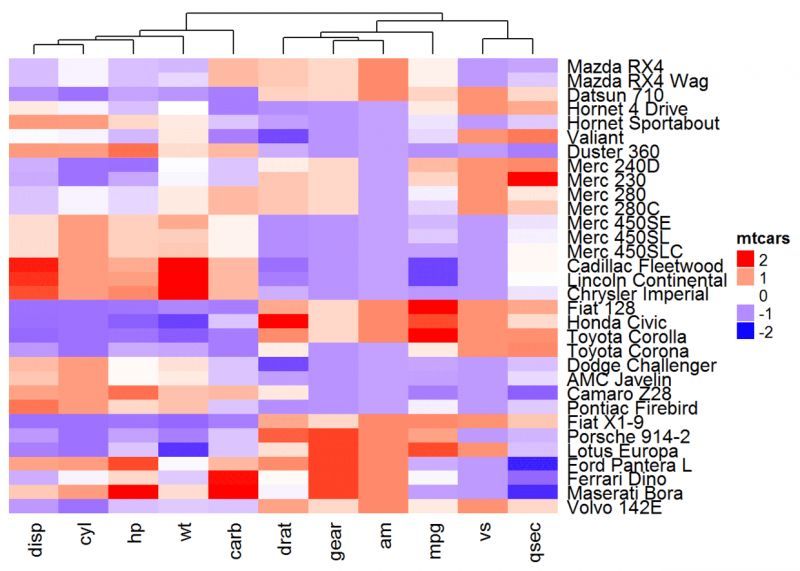
カラム群の高さや幅を変更するには、オプション column_dend_height を使用します。
と row_dend_width を指定します。
Heatmap(df, name = "mtcars", col = mycol, column_dend_height = unit(2, "cm")),
row_dend_width = unit(2, "cm") ) です。
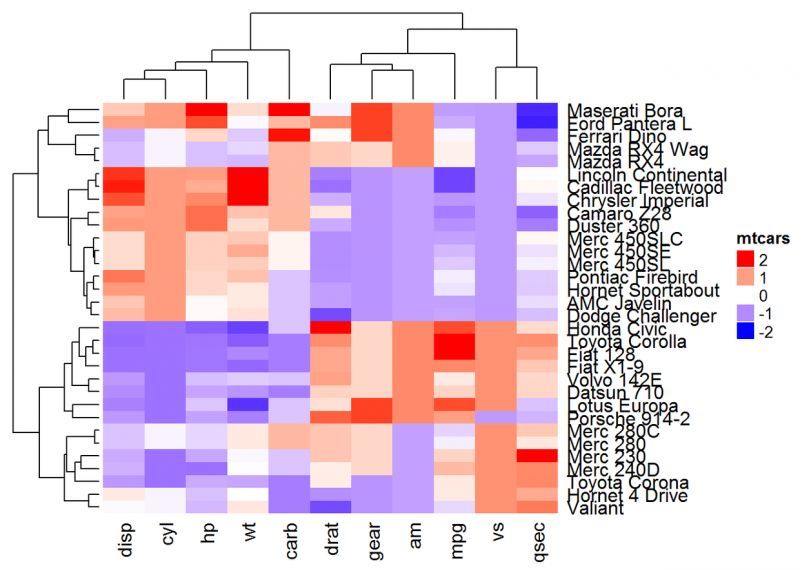
また、color_branches() を使用して、ツリーの外観をカスタマイズすることもできます。
ライブラリ(dendextend)
row_dend = hclust(dist(df)) # 行のクラスタリング
col_dend = hclust(dist(t(df))) # 列のクラスタリング
ヒートマップ(df, name = "mtcars", col = mycol, cluster_rows =)
color_branches(row_dend, k = 4), cluster_columns = color_branches(col_dend, k = 2))
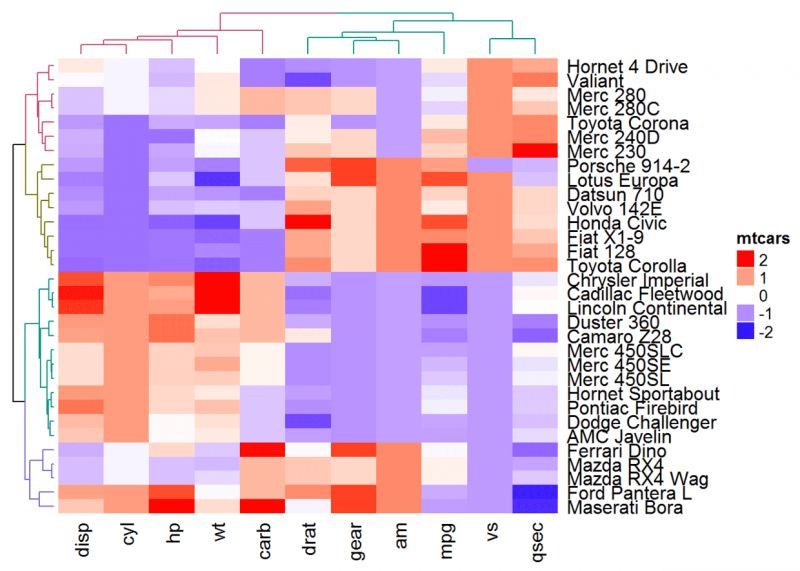
<スパン
クラスタリング距離のさまざまな計算方法
<スパン パラメータです。
clustering_distance_rows および clustering_distance_columns
行と列のクラスタリングにそれぞれメトリックを指定します。許容される値は "euclidean"、"max"、"manhattan"、"canberra"、"binary"、"minkowski" "pearson" "spearman" および "kendall"です。
Heatmap(df, name = "mtcars", clustering_distance_rows = "pearson",
clustering_distance_columns = "pearson")
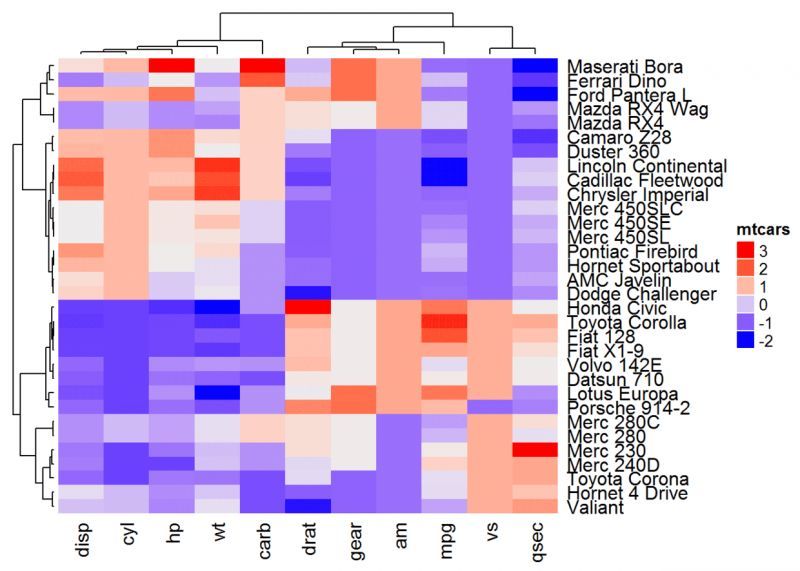
# 距離の計算をカスタマイズすることもできます
ヒートマップ(df, name = "mtcars", clustering_distance_rows = function(m) dist(m)))
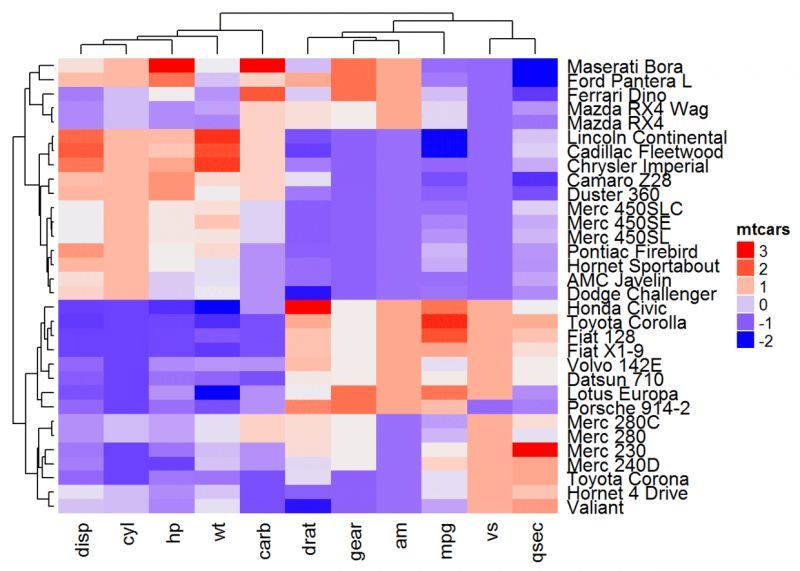
ヒートマップ(df, name = "mtcars", clustering_distance_rows = function(x, y) 1 - cor(x, y))
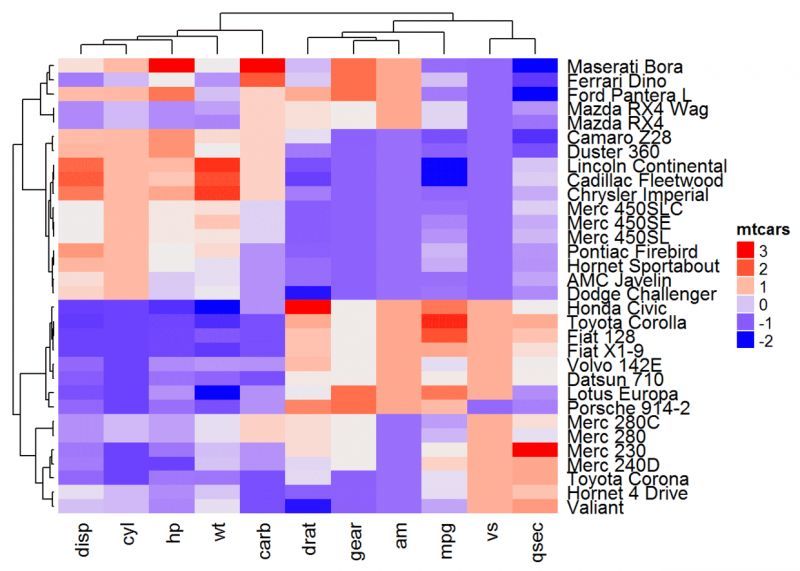
上記のRコードでは、通常、行のクラスタリングのメトリックを指定するパラメータclustering_distance_rowsの例を示していることに注意してください。パラメータ clustering_distance_columns (列クラスタリングのメトリック) には、同じメトリックを使用することが推奨されます。
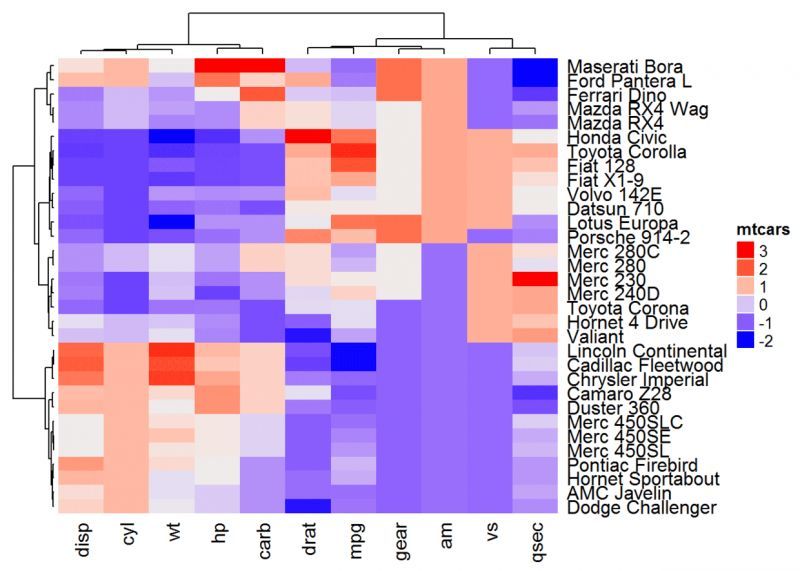
# クラスタリングメトリック関数
robust_dist = function(x, y) {. <未定義
qx = quantile(x, c(0.1, 0.9)) qy = quantile(y, c(0.1, 0.9)) l = x > qx[1] & x < qx[2] & y
> qy[1] & y < qy[2] x = x[l] y = y[l] sqrt(sum((x - y)^2)))}.
# ヒートマップ
Heatmap(df, name = "mtcars", clustering_distance_rows = robust_dist,
clustering_distance_columns = robust_dist。
col = colorRamp2(c(-2, 0, 2), c("purple", "white", "orange")) ))
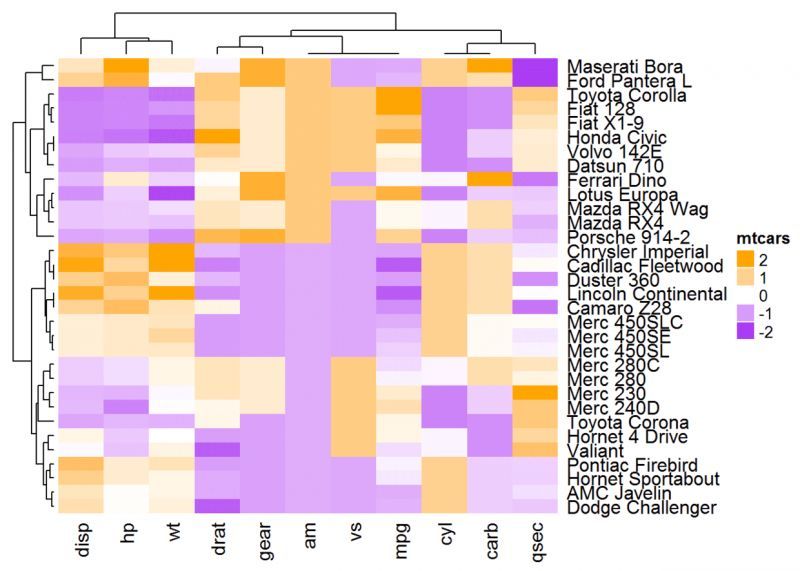
<スパン クラスタリング手法
<スパン パラメータです。
clustering_method_rows と clustering_method_columns を用いて、 階層型クラスタリングを行う方法を指定することができる。許容される値は hclust() 関数でサポートされているもので、次のようなものがあります。
"ward.D2"、"single"。 <スパン "complete"、その。 <スパン "平均", ... (hclustを参照)。
<ブロッククオートHeatmap(df, name = "mtcars", clustering_method_rows = "ward.D",
clustering_method_columns = "ward.D")
<スパン
ヒートマップ分割
<スパン
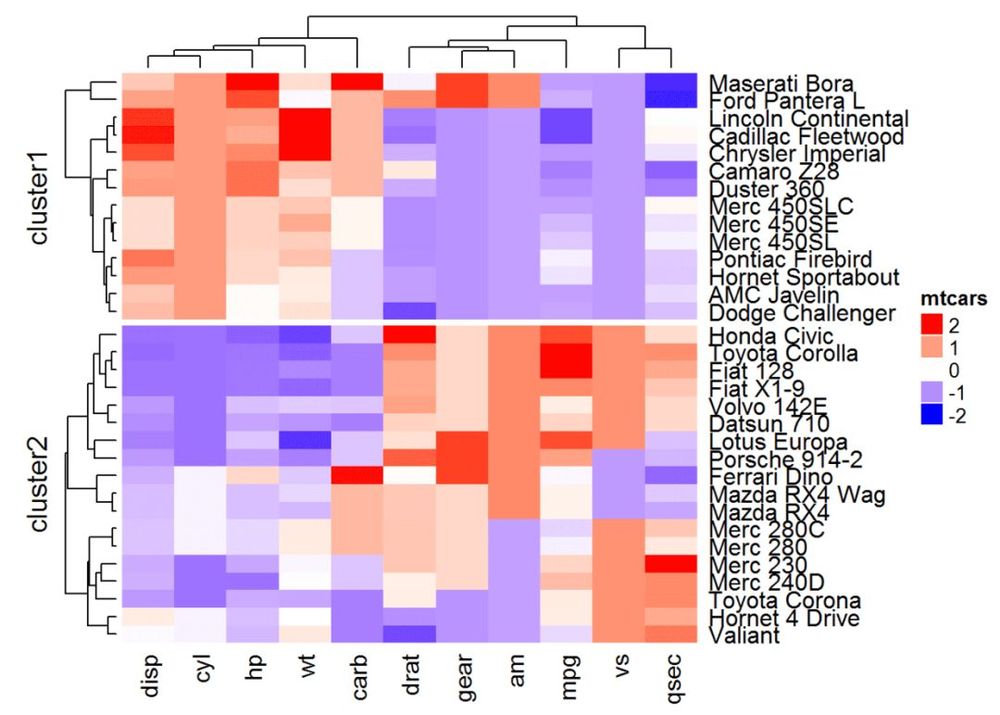
ヒートマップを分割する方法はたくさんあります。一つの解決策は、パラメトリックkmを使ったk-meansを適用することである。
k-meansを実行する際には、結果を後で正確に再現できるように、set.seed()関数を使用することが重要である
set.seed(1122)
# 2つのグループに分割Heatmap(df, name = "mtcars", col = mycol, k = 2)
# 行のクラスを指定するベクトルによって分割されます。
ヒートマップ(df, name = "mtcars", col = mycol, split = mtcars$cyl )
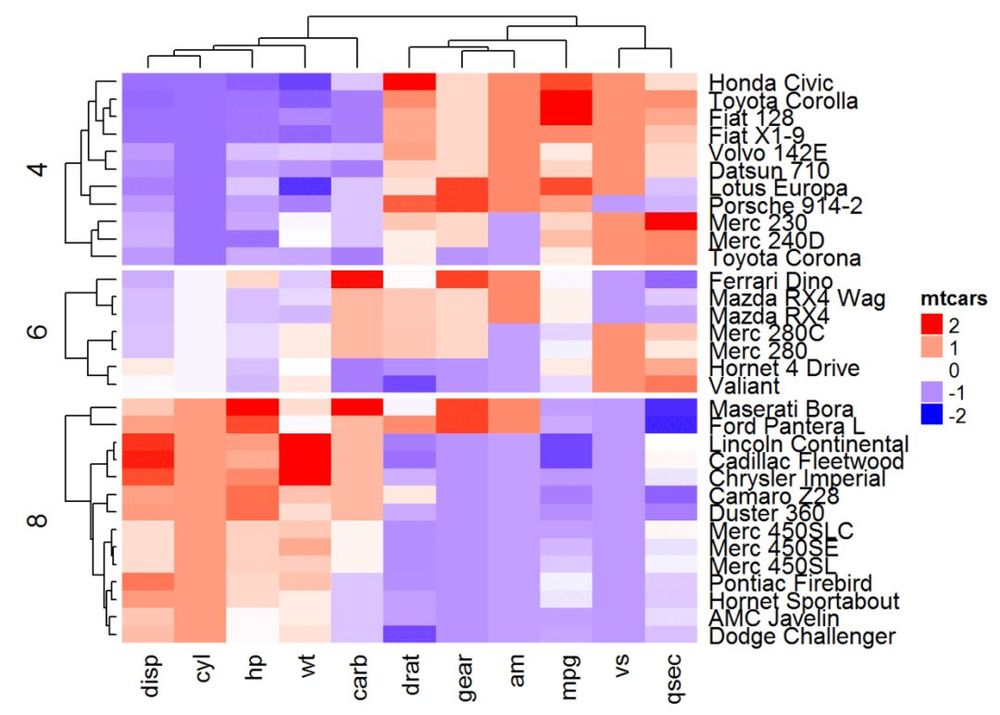
また、#splitは、異なるレベルの組み合わせでヒートマップの行を分割するデータボックスとすることもできます。
複数の変数を組み合わせて#split
ヒートマップ(df, name ="mtcars", col = mycol, split = data.frame(cyl = mtcars$cyl, am = mtcars$am))
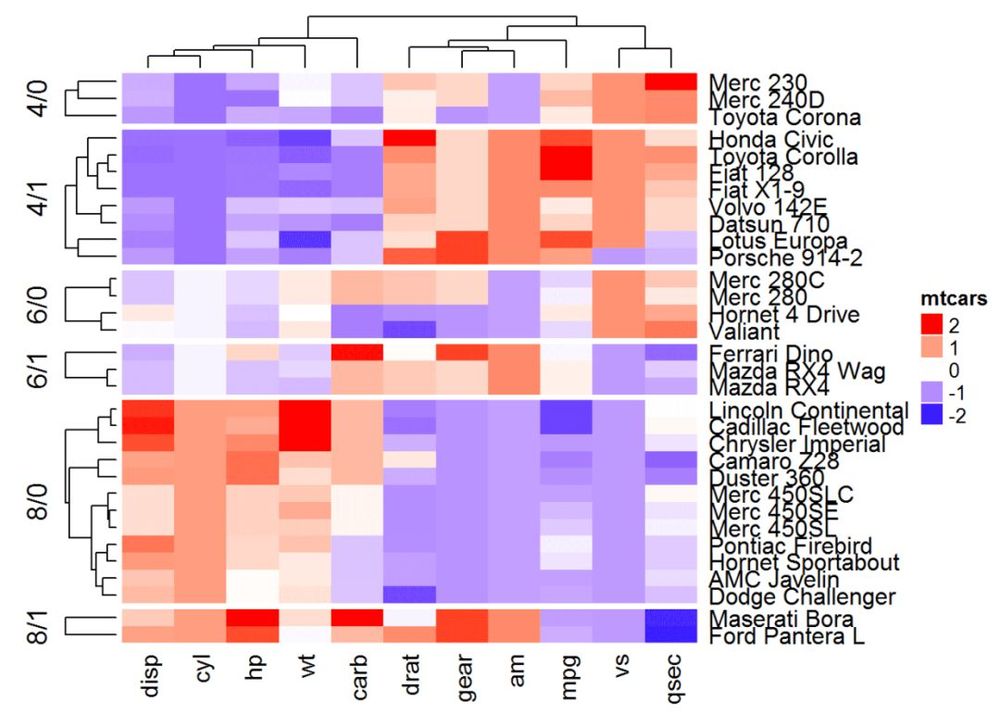
# kmと分割を組み合わせる
ヒートマップ(df, name ="mtcars", col = mycol, km = 2, split = mtcars$cyl)
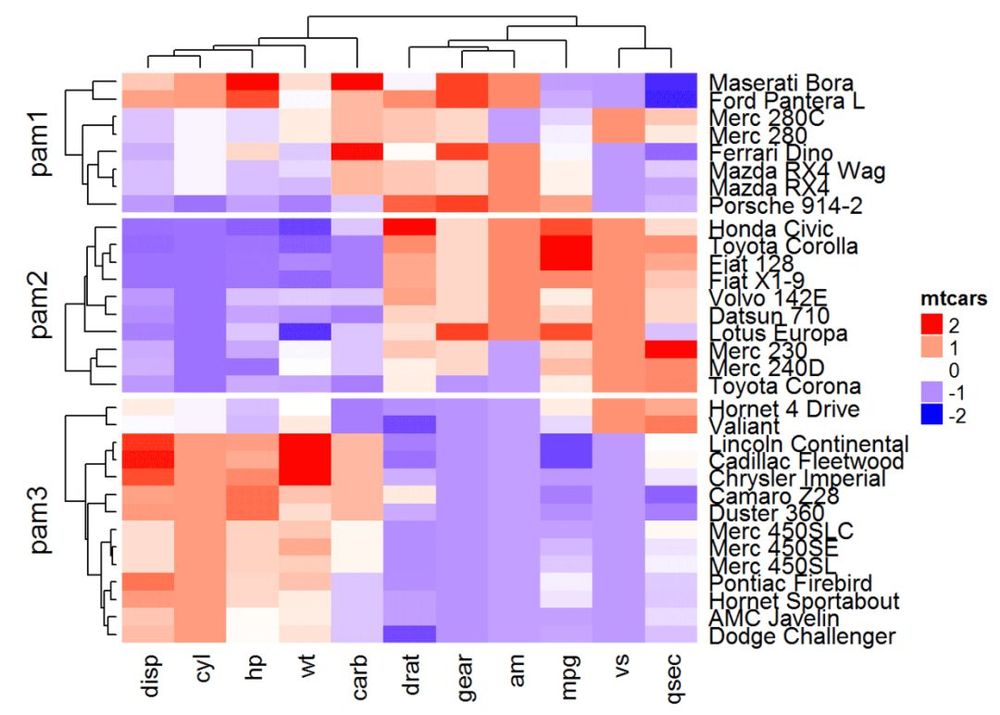
#分割のカスタマイズも可能
ライブラリ("cluster")
set.seed(1122)
pa = pam(df, k = 3)Heatmap(df, name = "mtcars", col = mycol, split = paste0("pam".Df, name = "mtcars", col = mycol, split = paste0("pam".Df)),
pa$clustering))
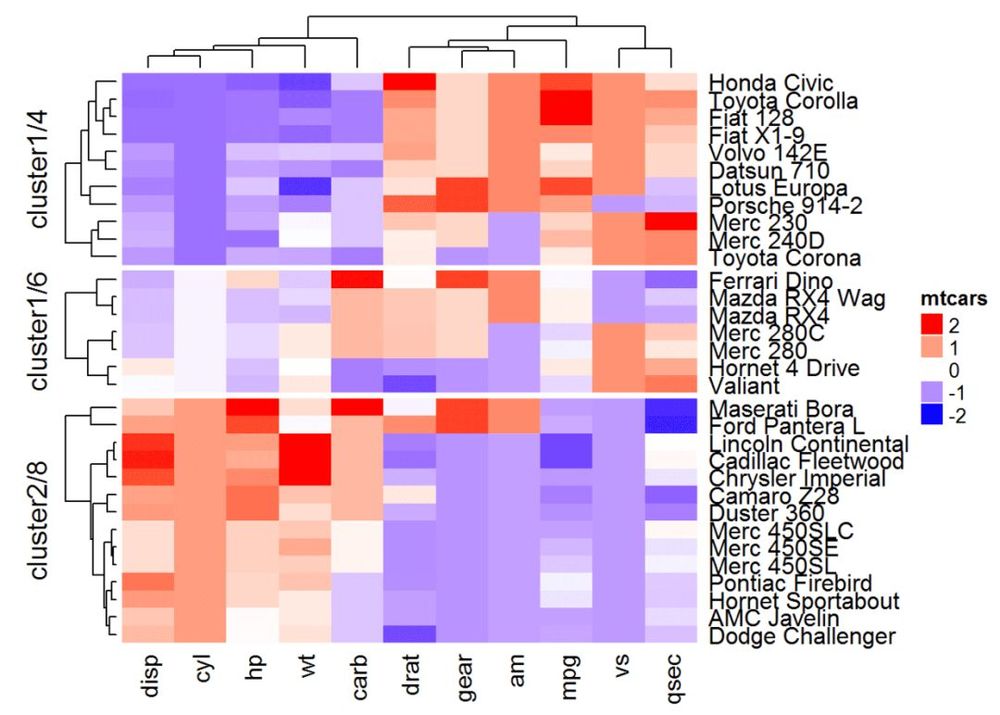
また、ユーザー定義のツリーと分割を組み合わせることも可能である。この場合、分割は単一の数値として指定することができる。
row_dend = hclust(dist(df)) # 行のクラスタリング
grow_dend = color_branches(row_dend, k = 4)
ヒートマップ(df, name = "mtcars", col = mycol, cluster_rows = row_dend, split = 2)
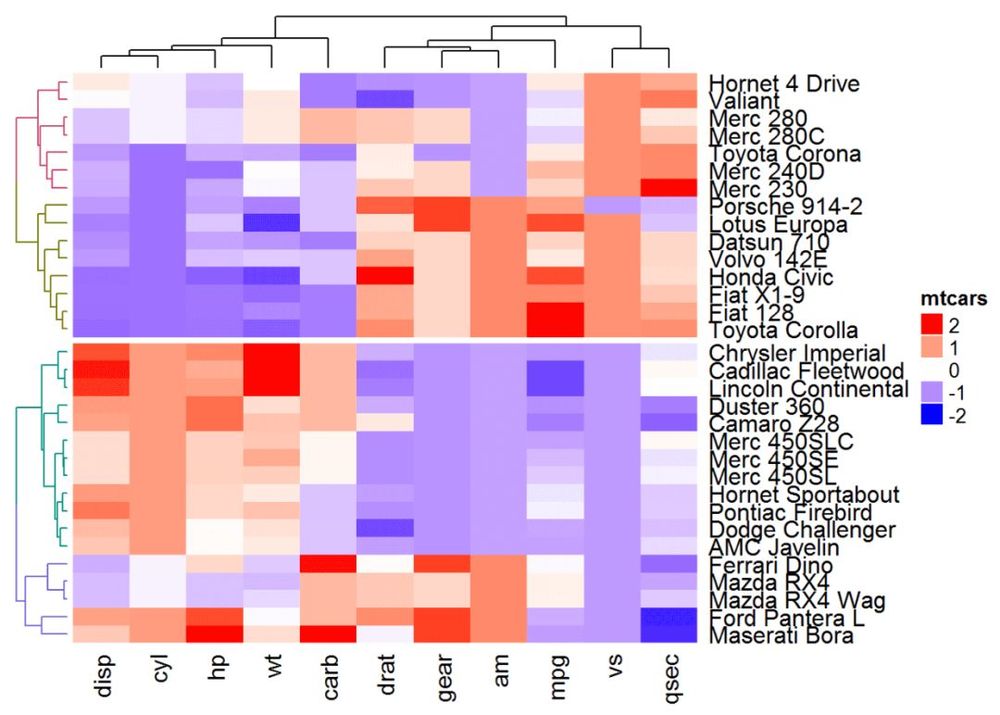
<スパン ヒートマップアノテーション
HeatmapAnnotation()で行や列の注釈を付けます。書式は ヒートマップアノテーション(df, name, col, show_legend)
df: 列名を含むdata.frame
name: ヒートマップアノテーションの名前
col: dfのカラムにマッピングされた色のリスト
# Transposedf <- t(df)
# 転置されたデータのヒートマップ
ヒートマップ(df, name ="mtcars", col = mycol)
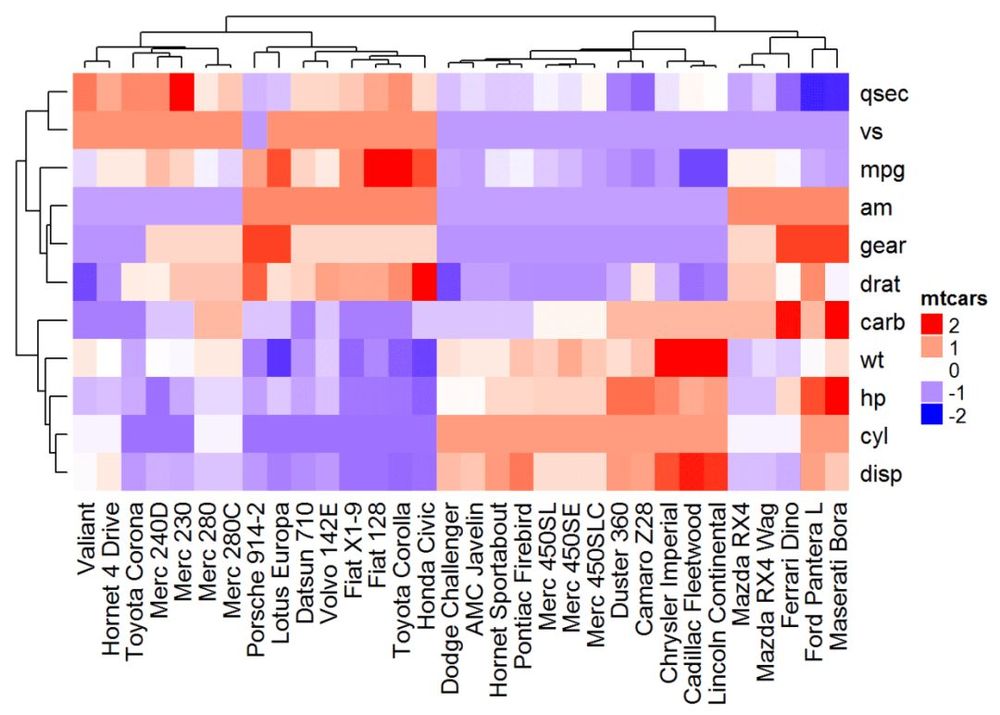
# アノテーションデータフレーム
annot_df <- data.frame(cyl = mtcars$cyl, am = mtcars$am, mpg = mtcars$mpg)
# 定性的変数の各レベルに色を定義する
# 連続変数 (mpg) のグラデーションカラーを定義する
col = list(cyl = c("4" = "green", "6" = "gray", "8" = "darkred"), am = c("0" ; = "yellow".JP), col = c(" 3" = " 5" ),
"1" = "orange"), mpg = colorRamp2(c(17, 25), c("lightblue", "purple")) )
# ヒートマップアノテーションの作成
ha <- HeatmapAnnotation(annot_df, col = col)
# ヒートマップとアノテーションを結合する
ヒートマップ(df, name = "mtcars", col = mycol, top_annotation = ha)
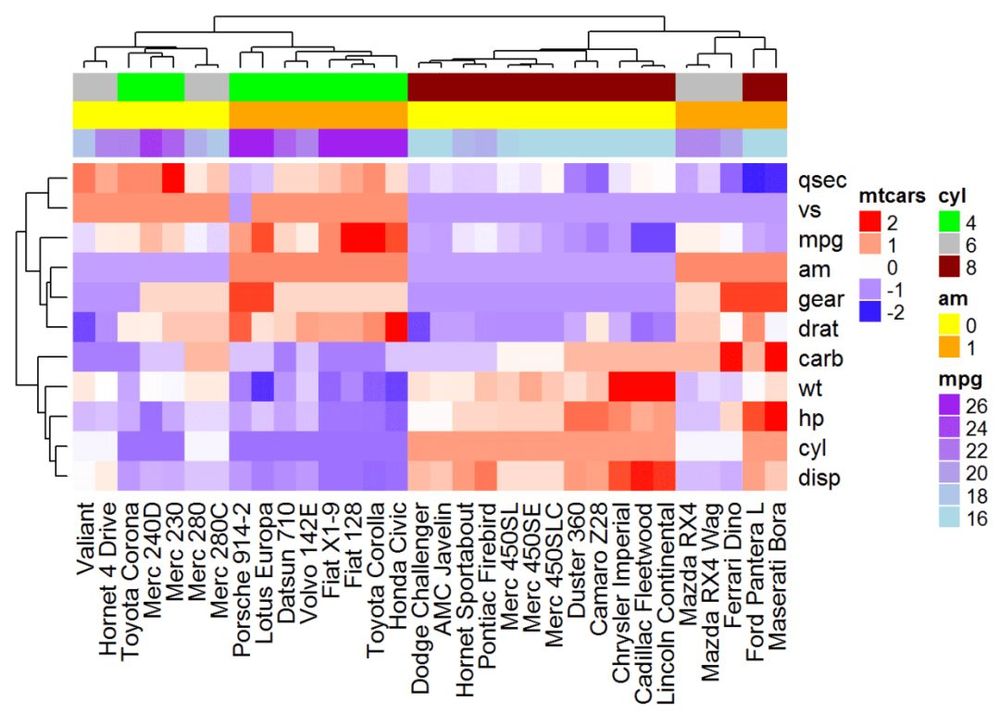
# show_legend = FALSE パラメータを使用すると、アノテーションの凡例を隠すことができます。
ha <- HeatmapAnnotation(annot_df, col = col, show_legend = FALSE)
ヒートマップ(df, name = "mtcars", col = mycol, top_annotation = ha)
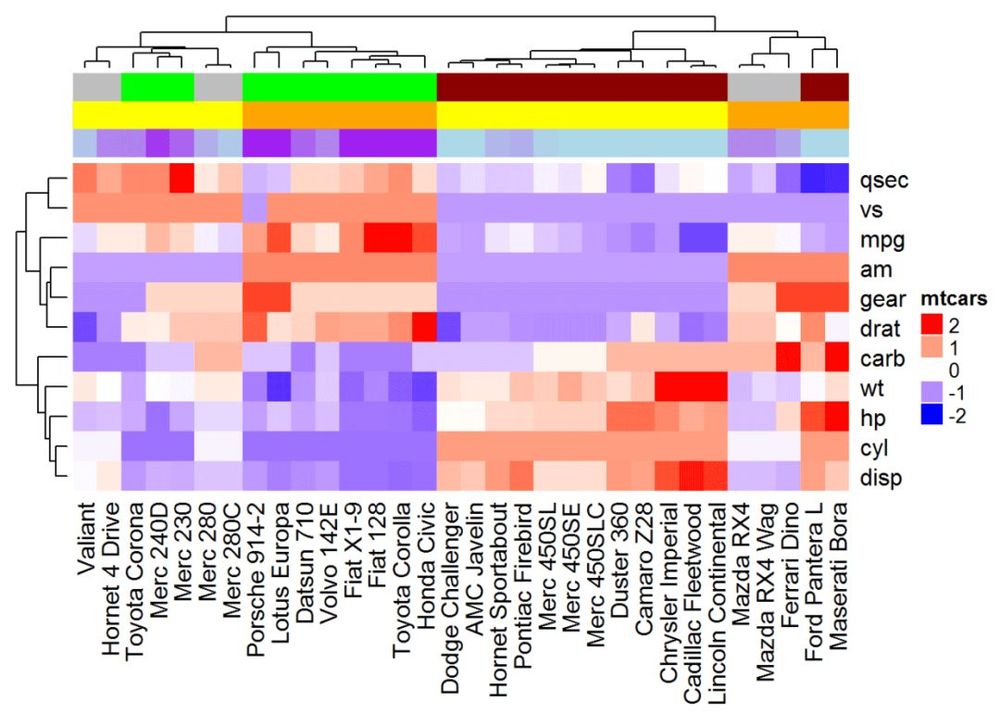
#コメント名は、以下のRコードで追加できます。
ライブラリ("GetoptLong")
# ヒートマップとアノテーションを合成する
ha <- HeatmapAnnotation(annot_df, col = col, show_legend = FALSE)
ヒートマップ(df, name = "mtcars", col = mycol, top_annotation = ha)
# 右側にアノテーション名を追加
for(an in colnames(annot_df)) { <未定義
seekViewport(qq("annotation_@{an}"))
grid.text(an, unit(1, "npc") + unit(2, "mm"), 0.5, default.units = "npc", just = "left")} となります。
# 左側にコメント名を追加するには、以下のコードを使用します。
# 左側にアノテーション名
for(an in colnames(annot_df)) { seekViewport(qq("annotation_@{an}")) grid.text(an,
unit(1, "npc") - unit(2, "mm"), 0.5, default.units = "npc", just = "left")} に変更しました。

<スパン 複雑なアノテーション
anno_point(),anno_barplot() を用いて、いくつかの基本的なグラフィックスでヒートマップに注釈を付ける。
anno_boxplot()、 anno_density() および anno_histogram() を使用します。
# 列の分布を表示するためのグラフィックをいくつか定義する
.hist = anno_histogram(df, gp = gpar(fill = "lightblue"))です.
.density = anno_density(df, type = "line", gp = gpar(col = "blue"))である。
ha_mix_top = HeatmapAnnotation(hist = .hist, density = .density)
# 行の分布を表示するためのグラフィックを定義する
.violin = anno_density(df, type = "violin", gp = gpar(fill = "lightblue"), which = "行")です。
.boxplot = anno_boxplot(df, which = "row")です。
ha_mix_right = HeatmapAnnotation(violin = .violin, bxplt = .boxplot, which = "row")。
幅 = 単位(4, "cm"))
# アノテーションとヒートマップを合成する
Heatmap(df, name = "mtcars", col = mycol, column_names_gp = gpar(fontsize = 8)),
top_annotation = ha_mix_top, top_annotation_height = unit(4, "cm"))) + ha_mix_right
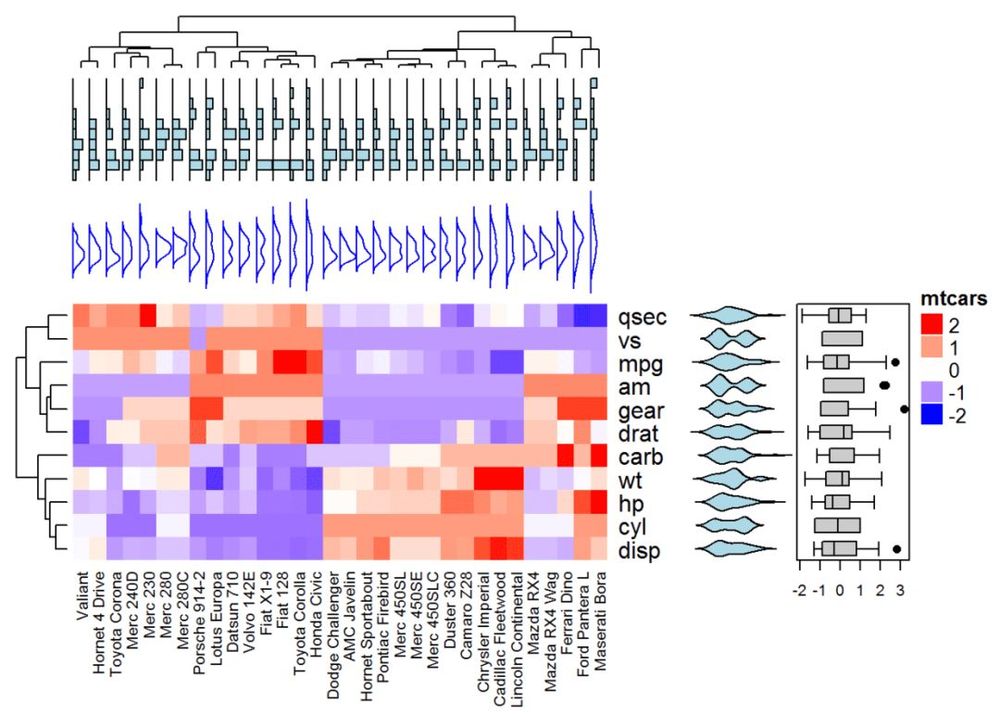
<スパン ヒートマップポートフォリオ
<スパン
#ヒートマップ1
ht1 = Heatmap(df, name = "ht1", col = mycol, km = 2, column_names_gp = gpar(fontsize = 9))
# ヒートマップ2
ht2 = Heatmap(df, name = "ht2", col = colorRamp2(c(-2, 0, 2), c("green", "white", "red"))), column_names_ gp = gpar(fontsize = 9)))
# 2つのヒートマップを合成する
ht1 + ht2
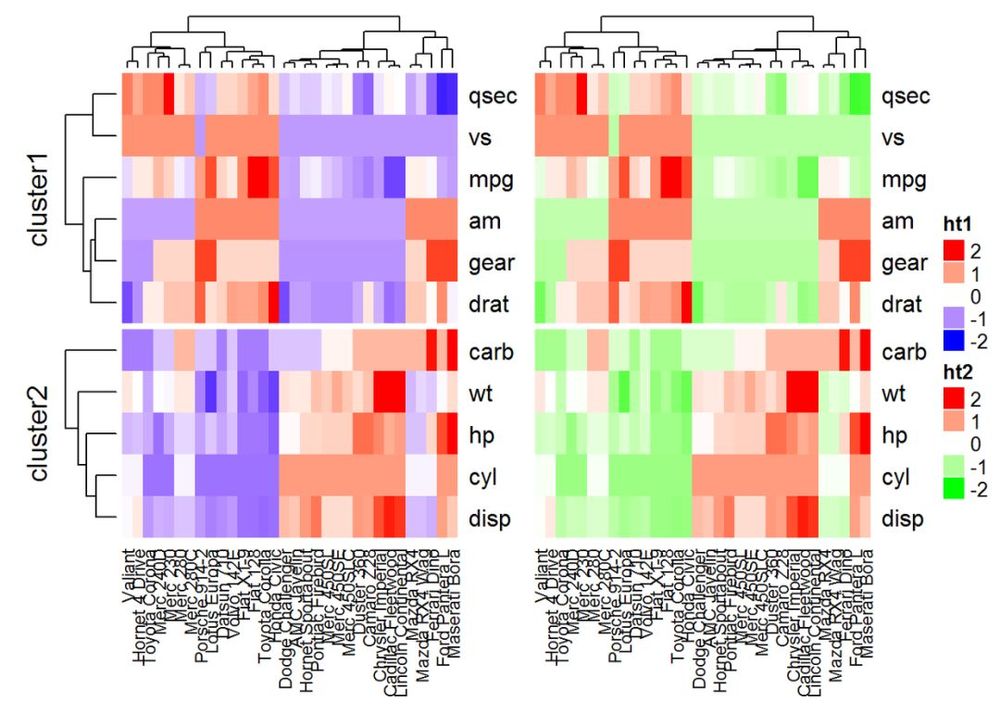
width = unit(3, "cm")) というオプションで、ヒートマップの大きさを制御することができます。複数のヒートマップを組み合わせる場合、最初のヒートマップがメインヒートマップとして扱われることに注意してください。残りのヒートマップのいくつかの設定は、メインヒートマップの設定に従って自動的に調整されます。これらの設定には、行クラスタやヘッダの削除、分割の追加などがあります。
draw(ht1 + ht2,
# タイトル
row_title = "2つのヒートマップ、行のタイトル"。
row_title_gp = gpar(col = "red")とします。
column_title = "2つのヒートマップ、列のタイトル"。
column_title_side = "bottom"です。
# ヒートマップ間のギャップ
ギャップ = ユニット(0.5, "cm"))
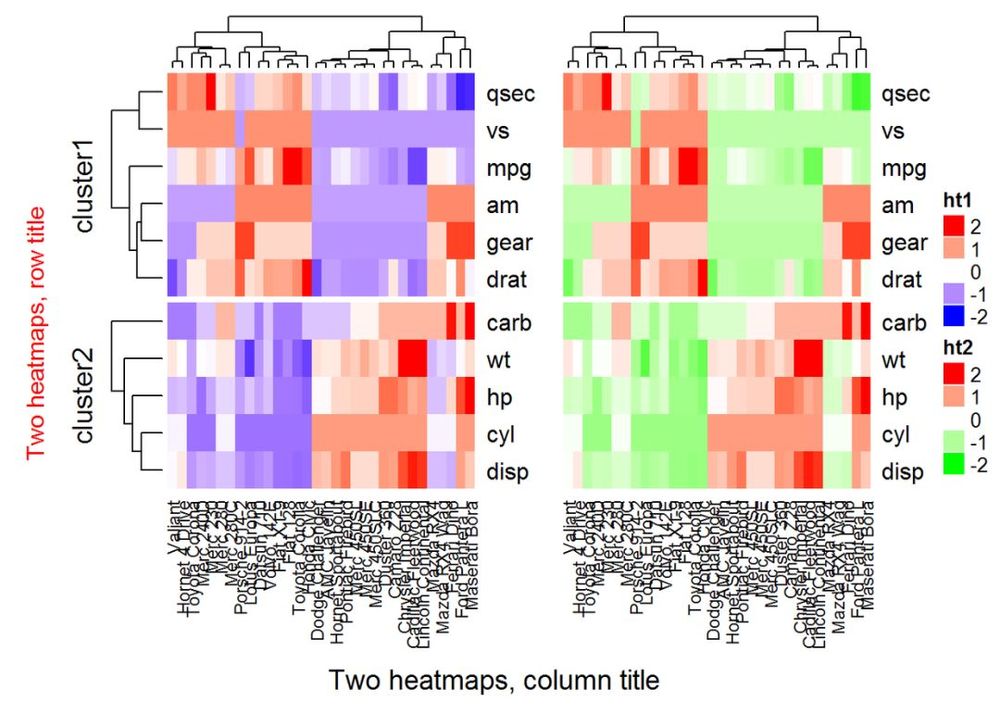
パラメータ show_heatmap_legend = FALSE, show_annotation_legend = FALSE を使用すると、凡例を削除することができます。
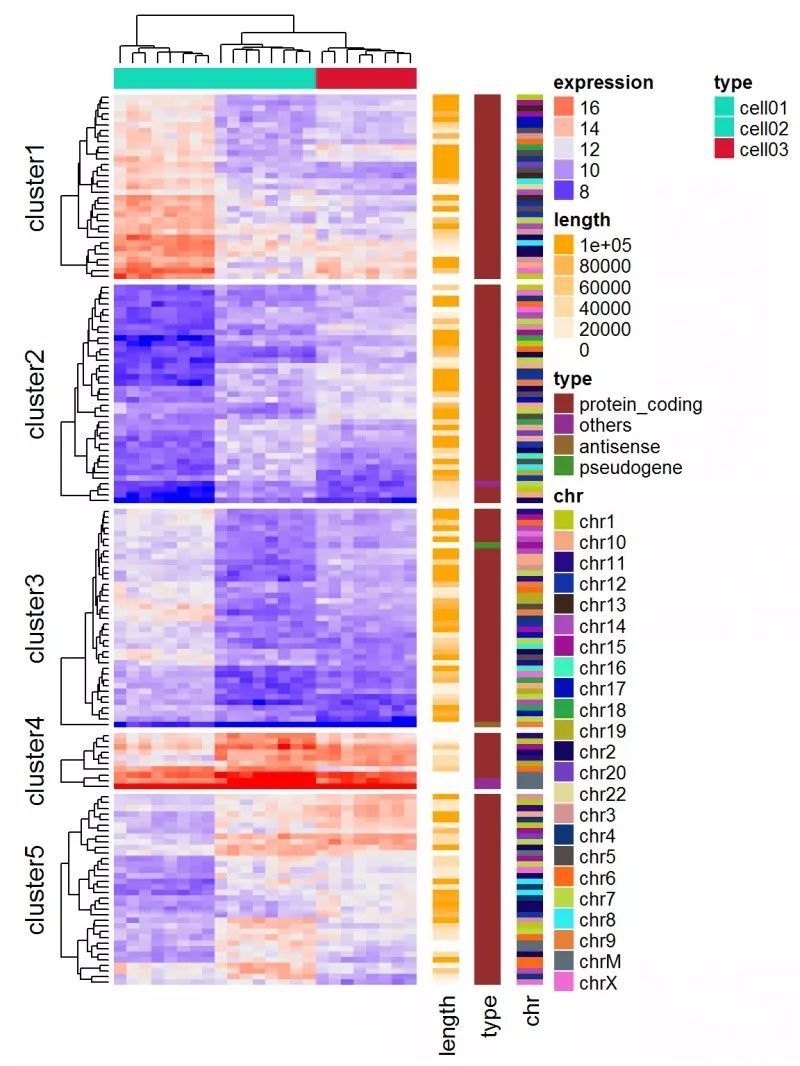
<スパン 遺伝子発現マトリックス
<スパン 遺伝子発現データでは、行が遺伝子、列がサンプル値を表す。発現ヒートマップの後に、遺伝子長や遺伝子型など、遺伝子に関する詳細な情報を付加することができます。
expr = readRDS(paste0(system.file(package = "ComplexHeatmap"), "/extdata/gene_expression.rds")))
mat = as.matrix(expr[, grep("cell", colnames(expr))])
type = gsub("sèmed+_", "", colnames(mat))
ha = HeatmapAnnotation(df = data.frame(type = type))
Heatmap(mat, name = "expression", km = 5, top_annotation = ha, top_annotation_height = unit(4, "mm")) を参照してください。
show_row_names = FALSE, show_column_names = FALSE) +。
Heatmap(expr$length, name = "length", width = unit(5, "mm"), col = colorRamp2(c(0, 100000), c("white", "orange")))... +
ヒートマップ(expr$type, name = "type", width = unit(5, "mm")) +
ヒートマップ(expr$chr, name = "chr", width = unit(5, "mm"), col = rand_color(length(unique(expr$chr))))
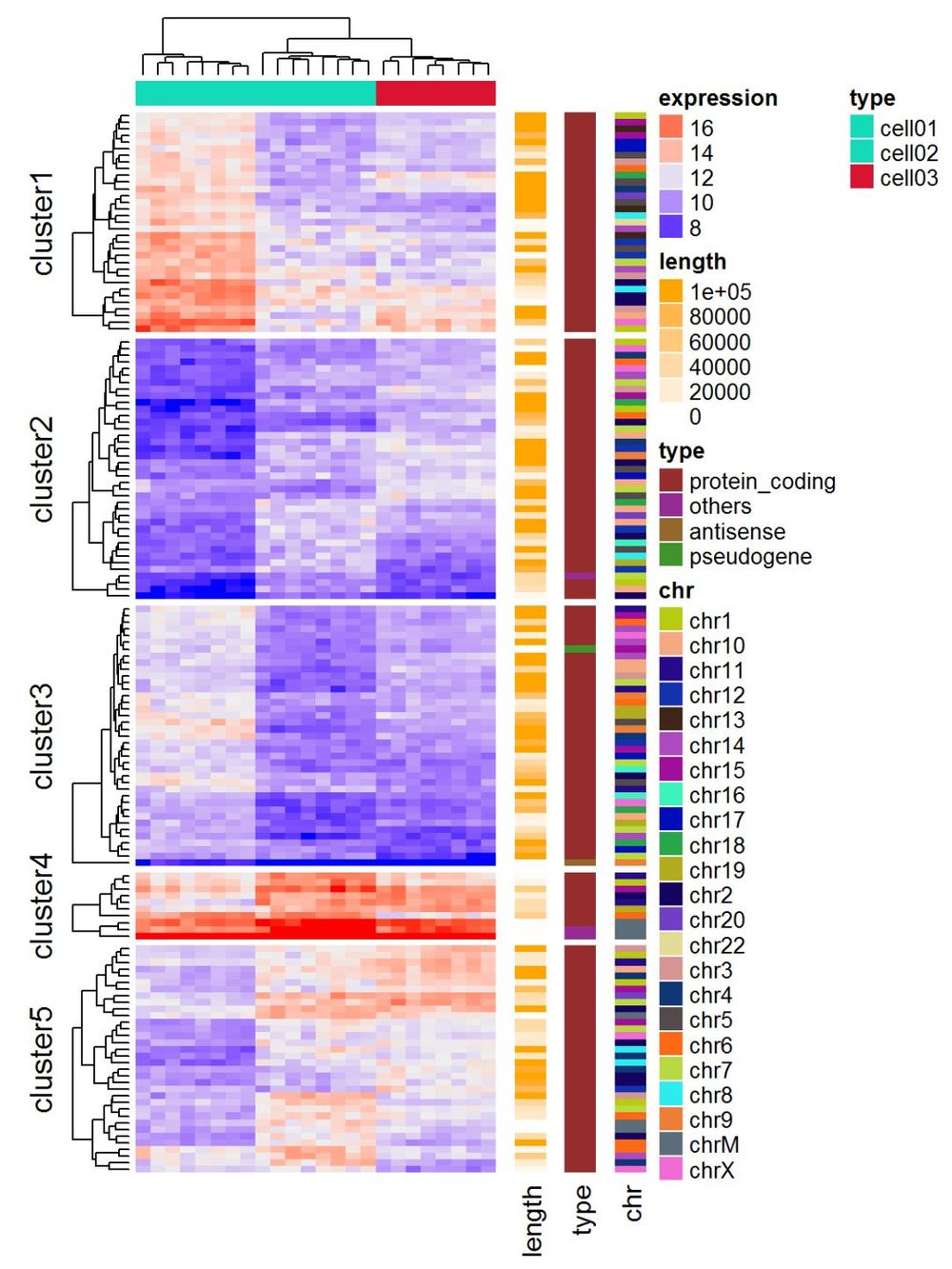
また、ゲノムの変化を可視化し、異なる分子レベル(遺伝子発現、DNAメチル化、...)を統合することが可能である。
<スパン
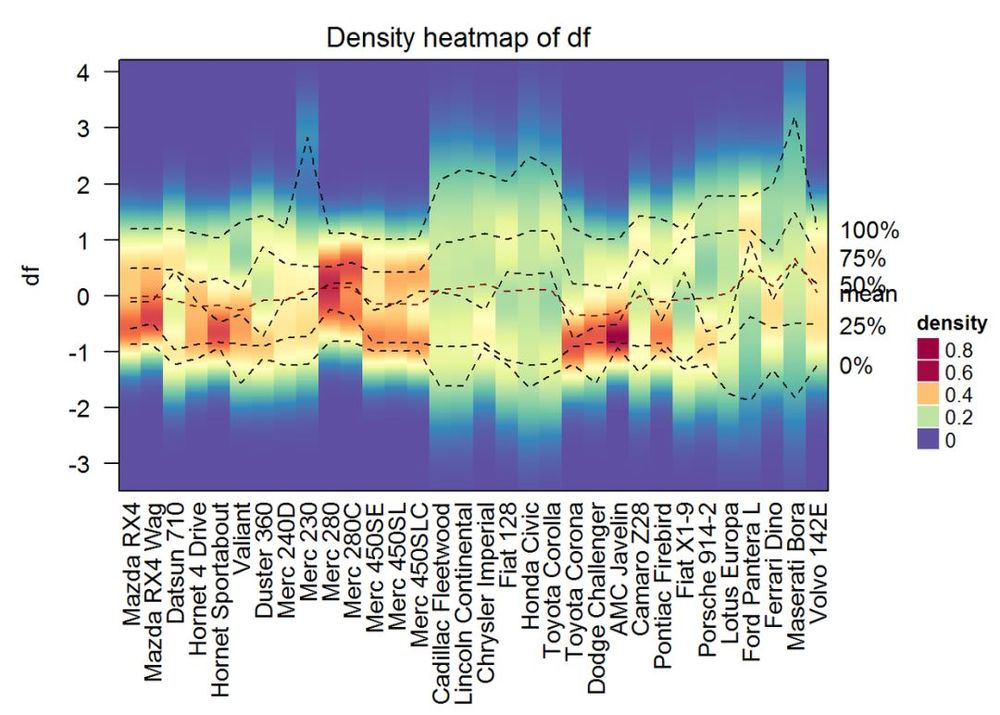
行列の列の分布を可視化する
関数densityHeatmap()を使用します。
密度ヒートマップ(df)
<スパン インフォメーション
sessionInfo()
## R バージョン 3.3.3 (2017-03-06)
## プラットフォーム:x86_64-w64-mingw32/x64 (64ビット)
## Running under: Windows 8.1 x64 (ビルド 9600)##で動作しています。
## ロケール
## [1] LC_COLLATE=中国語(簡体字)_中国.936
## [2] LC_CTYPE=中国語(簡体字)_中国.936
## [3] LC_MONETARY=中国語(簡体字)_中国.936
## [4] LC_NUMERIC=C
## [5] LC_TIME=中国語(簡体字)_中国.936 ##
## 添付の基本パッケージ
## [1] グリッド 統計 グラフィックス grDevices ユーティリティ データセット メソッド
## [8] ベース
##
## その他の付属パッケージ
## [1] GetoptLong_0.1.6 cluster_2.0.5 circlize_0.3.10
## [4] ComplexHeatmap_1.12.0 dendextend_1.4.0 d3heatmap_0.6.1.1
##[7] gplots_3.0.1 RColorBrewer_1.1-2
##
## 名前空間を介して読み込まれる(そして添付されない)。
## [1] Rcpp_0.12.9 DEoptimR_1.0-8 plyr_1.8.4
## [4] viridis_0.3.4 class_7.3-14 prabclus_2.2-6
## [7] bitops_1.0-6 base64enc_0.1-3 tools_3.3.3
## [10] digest_0.6.12 mclust_5.2.2 jsonlite_1.3
## [13] evaluate_0.10 tibble_1.2 gtable_0.2.0
## [16] lattice_0.20-34 png_0.1-7 yaml_2.1.14
## [19] mvtnorm_1.0-6 gridExtra_2.2.1 trimcluster_0.1-2
## [22] stringr_1.2.0 knitr_1.15.1 GlobalOptions_0.0.11
## [25] htmlwidgets_0.8 gtools_3.5.0 caTools_1.17.1
## [28] fpc_2.1-10 diptest_0.75-7 nnet_7.3-12
## [31] stats4_3.3.3 rprojroot_1.2 robustbase_0.92-7
## [34] flexmix_2.3-13 rmarkdown_1.3.9002 gdata_2.17.0
## [37] kernlab_0.9-25 ggplot2_2.2.1 magrittr_1.5
## [40] ウィスカー_0.3-2 バックポート_1.0.5 スケール_0.4.1
## [43] htmltools_0.3.5 modeltools_0.2-21 MASS_7.3-45
## [46] assertthat_0.1 shape_1.4.2 colorspace_1.3-2
## [49] KernSmooth_2.23-15 stringi_1.1.2 lazyeval_0.2.0
## [52] munsell_0.4.3 rjson_0.2.15

過去の記事
関連する行列可視化パッケージggcorrplotに関するR可視化ラーニングノート
ggplot2ラーニングノートシリーズ - ggplot2でエラーバーと有意性マーカーをプロットする
circos-plot を circlize パッケージでプロットする。
Rデータ操作学習ノート 変数の作成、名前の変更、データフュージョン
gganimate パッケージの R 可視化ラーニングノート
生物学者のためのRでプロットするためのレッスン02とamp;03
関連
-
不具合ログ】Prologでコンテンツが許可されていないエラー発生
-
eclipse Java ファイルオープン例外解決 : java.lang.StringIndexOutOfBoundsException: 文字列のインデックスが範囲外:26
-
画像ダウンロードの問題
-
python ランタイムプロンプト WebDriverException: メッセージ geckodriver' 実行ファイルが PATH にある必要があります。
-
C# 指定されたキーが辞書に存在しない。
-
java.security.cert.CertPathValidatorException を解決してください。認証パスのトラストアンカーが見つかりませんでした。
-
落とし穴を踏む-Uncaught Error: BootstrapのJavaScriptは、jQueryを必要とします。
-
Object reference not set to an instance of object "エラーの解決方法について。
-
ベクトル添え字の範囲外問題の解の1つ
-
Eclipseオンラインインストールエラー インストールするアイテムの収集中にエラーが発生しました。
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
C#のTask.Delay()とThread.Sleep()
-
要素 popover がクリックされると表示されない 問題が報告される 未定義のプロパティ '$refs' を読み取ることができない
-
CrtIsValidHeapPointerのエラーの解決法
-
mac git エラーを使用します。.git/FETCH_HEAD を開けない: パーミッションが拒否されました。
-
Gulpのマルチタスクエラーです。AssertionError [ERR_ASSERTION]: タスク関数を指定する必要があります ソリューション
-
raise NotImplementedError
-
MNISTの読み込みに失敗しました。[WinError 10060] コネクタがしばらくして正しく応答しなくなったため 解決策
-
ローカル変数sumが初期化されていない可能性がある
-
フロントエンドのhttpリクエストタイムアウトの概要について
-
AttributeError: module tensorflow no attribute app Solution