[解決済み] matplotlib で密度プロットを作成するには?
2022-03-06 01:53:13
質問
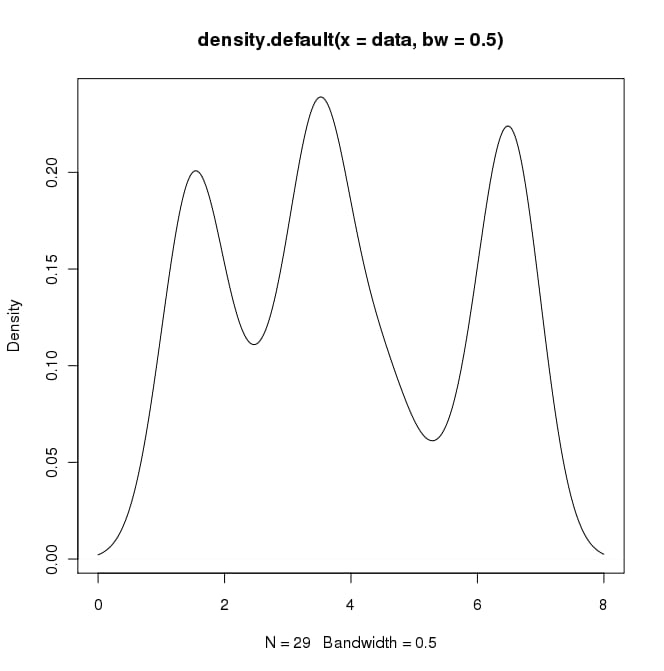
Rでは、次のようにすると目的の出力を作成することができます。
data = c(rep(1.5, 7), rep(2.5, 2), rep(3.5, 8),
rep(4.5, 3), rep(5.5, 1), rep(6.5, 8))
plot(density(data, bw=0.5))
<イグ
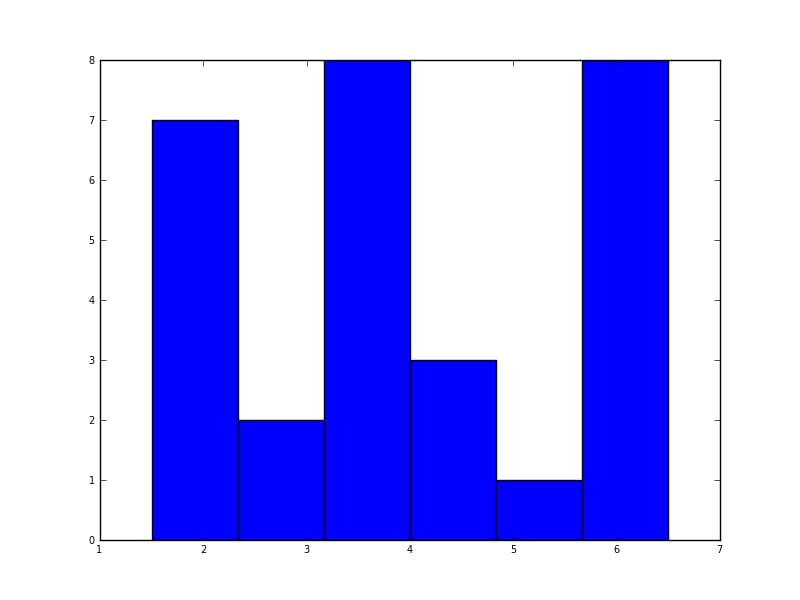
pythonでは(matplotlibで)最も近いのは単純なヒストグラムでした。
import matplotlib.pyplot as plt
data = [1.5]*7 + [2.5]*2 + [3.5]*8 + [4.5]*3 + [5.5]*1 + [6.5]*8
plt.hist(data, bins=6)
plt.show()
<イグ
また normed=True パラメータ が、ヒストグラムにガウシアンを当てはめようとする以外には、何も得られなかったのです。
私が最近試したのは、以下のあたりです。
scipy.stats
と
gaussian_kde
のように、Web上の例を参考にしましたが、今のところうまくいきません。
どのように解決するのですか?
Sven は、クラス
gaussian_kde
Scipyから生成されたものですが、Rで生成されたものとは全く違うことに気がつくでしょう。
gaussian_kde
は、帯域幅を自動的に推論しようとします。この帯域幅は、関数
covariance_factor
の
gaussian_kde
クラスがあります。まず、その関数を変更せずに得られるものは以下の通りです。
しかし、次のようなコードにすると
import matplotlib.pyplot as plt
import numpy as np
from scipy.stats import gaussian_kde
data = [1.5]*7 + [2.5]*2 + [3.5]*8 + [4.5]*3 + [5.5]*1 + [6.5]*8
density = gaussian_kde(data)
xs = np.linspace(0,8,200)
density.covariance_factor = lambda : .25
density._compute_covariance()
plt.plot(xs,density(xs))
plt.show()
私は
というのは、Rから得られるものにかなり近いと思います。
gaussian_kde
は変更可能な関数を使用しています。
covariance_factor
を使用して帯域幅を計算しています。関数を変更する前、このデータに対してcovariance_factorが返した値は約0.5であった。これを下げると帯域幅が狭くなりました。私は
_compute_covariance
この関数を変更した後、すべてのファクターが正しく計算されるようにしました。Rのbwパラメータと正確に対応しているわけではありませんが、正しい方向に向かうための助けになれば幸いです。
関連
-
PythonによるLeNetネットワークモデルの学習と予測
-
[解決済み】 AttributeError("'str' object has no attribute 'read'")
-
[解決済み] プログラムの実行やシステムコマンドの呼び出しはどのように行うのですか?
-
[解決済み] リストのリストからフラットなリストを作るには?
-
[解決済み] Matplotlibでプロットを表示するのではなく、画像ファイルに保存する。
-
[解決済み] Matplotlib で凡例をプロットの外側に配置する方法
-
[解決済み] IPythonノートブックmatplotlibプロットをインラインで行う方法
-
[解決済み】ネストされたディレクトリを安全に作成するには?
-
[解決済み] matplotlib のプロットでフォントサイズを変更する方法
-
[解決済み】2つの辞書を1つの式でマージする(辞書の和をとる)には?)
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
ピローによる動的キャプチャ認識のためのPythonサンプルコード
-
Python jiabaライブラリの使用方法について説明
-
Python 可視化 big_screen ライブラリ サンプル 詳細
-
[解決済み] _tkinter.TclError: 表示名がなく、$DISPLAY環境変数もない。
-
[解決済み】Python regex AttributeError: 'NoneType' オブジェクトに 'group' 属性がない。
-
[解決済み】ImportError: PILという名前のモジュールがない
-
[解決済み】syntaxError: 'continue' がループ内で適切に使用されていない
-
[解決済み】NameError: 名前 'self' が定義されていません。
-
[解決済み】Flaskのテンプレートが見つからない【重複あり
-
[解決済み】django インポートエラー - core.managementという名前のモジュールがない