[解決済み] パラメータが多すぎるクラス:より良い設計戦略?
2023-07-28 02:11:26
質問
私は神経細胞のモデルを扱っています。私が設計しているクラスの1つは、ニューロン(一緒に接続されたいくつかのコンパートメント)の位相的な記述であるセルクラスです。これは多くのパラメータを持っていますが、それらはすべて関連するものです。
軸索セグメントの数、頂点の二等分、体軸の長さ、体軸の直径、頂点の長さ、分岐のランダム性、分岐の長さなどなど...全部で15ほどのパラメータがあります!
これらをすべてデフォルト値に設定することもできますが、パラメータが何行もあるため、私のクラスはおかしなことになっています。このようなことは他の人にも時々起こることだと思いますが、これを設計するための何か明らかな良い方法があるのでしょうか、それとも私は正しいことをしているのでしょうか?
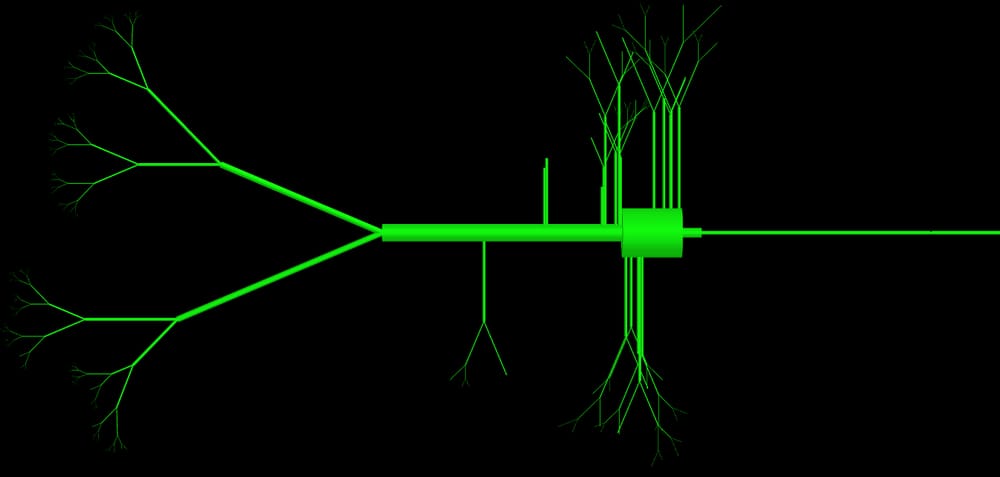
UPDATEです。 このクラスは膨大な数のパラメータ(>15)を持っていますが、それらはすべて使用され、細胞のトポロジーを定義するために必要です。問題は、このクラスが作成する物理的なオブジェクトが非常に複雑であることです。このクラスが生成するオブジェクトの画像表示を添付します。経験豊富なプログラマは、定義にこれほど多くのパラメータを避けるために、どのようにこれを違う方法で行うでしょうか?
class LayerV(__Cell):
def __init__(self,somatic_dendrites=10,oblique_dendrites=10,
somatic_bifibs=3,apical_bifibs=10,oblique_bifibs=3,
L_sigma=0.0,apical_branch_prob=1.0,
somatic_branch_prob=1.0,oblique_branch_prob=1.0,
soma_L=30,soma_d=25,axon_segs=5,myelin_L=100,
apical_sec1_L=200,oblique_sec1_L=40,somadend_sec1_L=60,
ldecf=0.98):
import random
import math
#make main the regions:
axon=Axon(n_axon_seg=axon_segs)
soma=Soma(diam=soma_d,length=soma_L)
main_apical_dendrite=DendriticTree(bifibs=
apical_bifibs,first_sec_L=apical_sec1_L,
L_sigma=L_sigma,L_decrease_factor=ldecf,
first_sec_d=9,branch_prob=apical_branch_prob)
#make the somatic denrites
somatic_dends=self.dendrite_list(num_dends=somatic_dendrites,
bifibs=somatic_bifibs,first_sec_L=somadend_sec1_L,
first_sec_d=1.5,L_sigma=L_sigma,
branch_prob=somatic_branch_prob,L_decrease_factor=ldecf)
#make oblique dendrites:
oblique_dends=self.dendrite_list(num_dends=oblique_dendrites,
bifibs=oblique_bifibs,first_sec_L=oblique_sec1_L,
first_sec_d=1.5,L_sigma=L_sigma,
branch_prob=oblique_branch_prob,L_decrease_factor=ldecf)
#connect axon to soma:
axon_section=axon.get_connecting_section()
self.soma_body=soma.body
soma.connect(axon_section,region_end=1)
#connect apical dendrite to soma:
apical_dendrite_firstsec=main_apical_dendrite.get_connecting_section()
soma.connect(apical_dendrite_firstsec,region_end=0)
#connect oblique dendrites to apical first section:
for dendrite in oblique_dends:
apical_location=math.exp(-5*random.random()) #for now connecting randomly but need to do this on some linspace
apsec=dendrite.get_connecting_section()
apsec.connect(apical_dendrite_firstsec,apical_location,0)
#connect dendrites to soma:
for dend in somatic_dends:
dendsec=dend.get_connecting_section()
soma.connect(dendsec,region_end=random.random()) #for now connecting randomly but need to do this on some linspace
#assign public sections
self.axon_iseg=axon.iseg
self.axon_hill=axon.hill
self.axon_nodes=axon.nodes
self.axon_myelin=axon.myelin
self.axon_sections=[axon.hill]+[axon.iseg]+axon.nodes+axon.myelin
self.soma_sections=[soma.body]
self.apical_dendrites=main_apical_dendrite.all_sections+self.seclist(oblique_dends)
self.somatic_dendrites=self.seclist(somatic_dends)
self.dendrites=self.apical_dendrites+self.somatic_dendrites
self.all_sections=self.axon_sections+[self.soma_sections]+self.dendrites
どのように解決するのですか?
UPDATEしてください。 この方法はあなたの特定のケースには適しているかもしれませんが、間違いなく欠点があります。 はアンチパターンなのか?
この方法を試してみてください。
class Neuron(object):
def __init__(self, **kwargs):
prop_defaults = {
"num_axon_segments": 0,
"apical_bifibrications": "fancy default",
...
}
for (prop, default) in prop_defaults.iteritems():
setattr(self, prop, kwargs.get(prop, default))
次に
Neuron
のようにします。
n = Neuron(apical_bifibrications="special value")
関連
-
[解決済み] パラメータを持つデコレータ?
-
[解決済み] matplotlib で多数のサブプロットでサブプロットサイズ/スペーシングを改善する
-
[解決済み] 値が多すぎて展開できない」、dictを反復する。 key=>string, value=>list
-
[解決済み】メソッドの型ヒントは、どのようにエンクロージャクラスの型を使用するのですか?
-
[解決済み】多重継承で親クラスの__init__を呼び出す、正しい方法は?
-
[解決済み] 開いている数字が多すぎる場合の警告
-
[解決済み] Pythonでクラスを設計するには?
-
[解決済み] googletransがエラー 'NoneType' オブジェクトに 'group' 属性がない、と言って動かなくなった。
-
[解決済み] virtualenv の `--no-site-packages` オプションを元に戻す。
-
[解決済み] djangoのQueryDictをPythonのDictに変更するには?
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
[解決済み] 名前付きタプルおよびオプションのキーワード引数のデフォルト値
-
[解決済み] PILからopenCVフォーマットへの変換
-
[解決済み] 辞書のキーと値を交換するにはどうすればよいですか?
-
[解決済み] データフレームをソートした後にインデックスを更新する
-
[解決済み] 値で列挙名を取得する [重複]。
-
[解決済み] Pythonでマルチプロセッシングキューを使うには?
-
[解決済み] subprocess.run()の出力を抑制またはキャプチャするには?
-
[解決済み] Flaskで非同期タスクを作る
-
[解決済み] 単純な文字列からtimedeltaオブジェクトを作成する方法
-
[解決済み] Python の sorted() はどのようなアルゴリズムを使っているのですか?重複